ポイント
- 新しい1細胞解析法scRepli-RamDA-seq(scRR-seq)を開発。この手法により、個々の細胞においてゲノムDNAと遺伝子発現を同時に解析できるようになった。
- scRR-seqは、DNAとRNAの双方について、高品質かつ高解像度の解析を実現。
- scRR-seqを用いることで、単一細胞内におけるDNAとRNAの直接的な関係がわかり、従来の手法では得られなかった知見を引き出すことが可能になった。
- scRR-seqは強力かつ汎用性の高いツールであり、疾患メカニズムの解明から胚発生研究まで幅広い研究分野で新たな発見をもたらすことが期待される。
概要
東京科学大学 総合研究院 難治疾患研究所の二階堂愛教授(理化学研究所チームディレクター)と林哲太郎連携研究員(理化学研究所技師)は、三重大学 生物資源学研究科の竹林慎一郎教授、プーンパーム・ラウィン助教(理化学研究所客員研究員)、大学院生(研究当時)の米田泰城さん、大学院生の今田泰斗さん、理化学研究所の平谷伊智朗チームディレクターらの研究グループと共同で、scRepli-RamDA-seq(scRR-seq)と呼ばれる新しい1細胞解析技術[用語1]を開発しました。この手法により、1つの細胞の中でゲノムDNA[用語2]とRNAの両方を高解像度で同時に解析することが可能となりました。DNAの変化と遺伝子発現の変化を直接的に結び付けることができるため、従来の技術ではアプローチの難しかった課題の解決につながることが期待されます。
近年、1細胞解析技術の進展は、細胞集団の解析では捉えられない細胞間の不均一性(細胞ごとの個性)を明らかにすることで生物学の様々な分野に革新を起こし、希少な異常細胞の同定など数多くの発見をもたらしてきました。しかし、既存の1細胞解析法の多くはDNAとRNAを別々に扱うため、これらの分子がどのように機能的に結び付いているかという理解には限界がありました。この課題を克服するために、共同研究グループがこれまでに確立してきた2つの最先端1細胞解析法、すなわちDNAコピー数を高解像度でシーケンス解析するscRepli-seqと、高感度のRNAシーケンス法であるRamDA-seqを組み合わせることで、1つの細胞からDNAとRNAを同時に解析できるようにscRR-seqを設計しました。scRR-seqを用いることで、新しい細胞周期進行マーカーを発見し、またDNAコピー数と遺伝子発現量の関係は必ずしも単純に正の相関を示すわけではないことも明らかになりました。
本研究は、科学雑誌「Nature Communications」(12月15日付)に掲載されました。
背景
これまで、DNAやRNAの研究の多くは多数の細胞をまとめて解析する方法で行われてきました。このアプローチは有用ではありますが、集団全体を平均化した情報しか得られず、個々の細胞の違い、いわゆる細胞の不均一性(図1a)を捉えることはできませんでした。近年、次世代シーケンス(NGS)[用語3]、単一細胞の分離、そして高感度の核酸増幅技術[用語4]といった新しい技術の登場により、DNAとRNAをたった1つの細胞レベルで研究できるようになりました。これによって、ゲノムDNAの働きや細胞ごとの遺伝子活性の違いをより明確に理解することが可能になっています。
竹林教授らの共同研究グループはこれまでに、動物細胞研究に利用できる2つの先進的な1細胞解析技術を開発してきました。
- scRepli-seq:個々の細胞から取り出した極微量のゲノムDNAを全ゲノム増幅(WGA)[用語5]により増やし、DNAコピー数[用語6]を高解像度で測定する技術(Takahashi et al., Nature Genetics 2019, Miura et al., Nature Protocols 2020)。DNAコピー数変異(CNV)[用語7]の検出やDNA複製[用語8]の状態を推定することができる(図1b)。
- RamDA-seq:非コードRNA[用語9]を含む全長RNA[用語10]を高感度で取得できる技術(Hayashi et al., Nature Communications 2018)(図1c)。
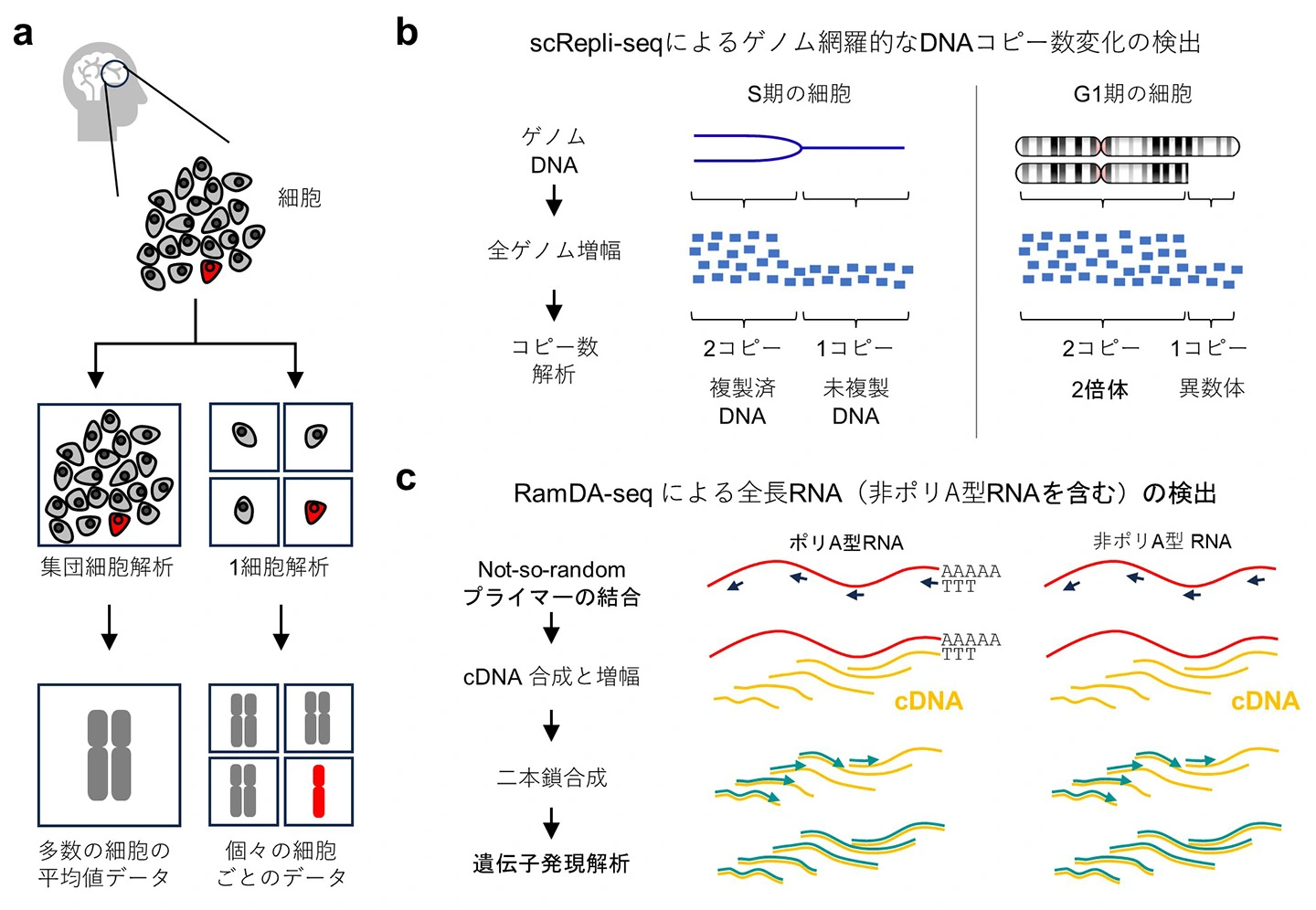
(a) 集団細胞の解析では細胞間の違いが平均化されて見えなくなるのに対し、1細胞解析は細胞ごとの多様性を直接捉えることができる。
(b) scRepli-seqは、1細胞に由来するごく微量のゲノムDNAを比較的均一に増幅し、DNAコピー数の解析を可能にする技術である。scRepli-seqでS期細胞を解析した場合は、コピー数からゲノムの複製状態を推定できる (2コピー = 複製済み; 1コピー = 未複製)。一方、G1期細胞などを解析した場合は、複製とは関係のないコピー数変動といったゲノム不安定化を検出できる。
(c) RamDA-seqは、「Not-so-random」プライマーと呼ばれる特殊なプライマー(矢印)を用いたcDNA合成を行い、さらに鎖置換増幅法を行うことで、高感度に1細胞由来のごく微量のRNAを検出する技術である。これにより、完全長RNAの解析が可能となり、ポリ(A)RNAに加えて従来法では捉えられない非ポリ(A)RNAも解析できる。
1細胞解析技術は非常に強力な実験手法ですが、DNAとRNAの両方を1つの細胞から同時に採取して調べない限り、ゲノムDNAと遺伝子活性がどのように連動しているかを明らかにすることはできません。これまでDNA/RNA同時解析法は報告されていましたが、感度や解像度が十分でなく、両者の関係を明確に示すことは困難でした。このような状況の中で、今回本共同研究グループは、これまでに独自に開発したscRepli-seqとRamDA-seqを組み合わせることで、新しい1細胞同時解析手法scRepli-RamDA-seq(scRR-seq)の開発に取り組みました。
研究成果
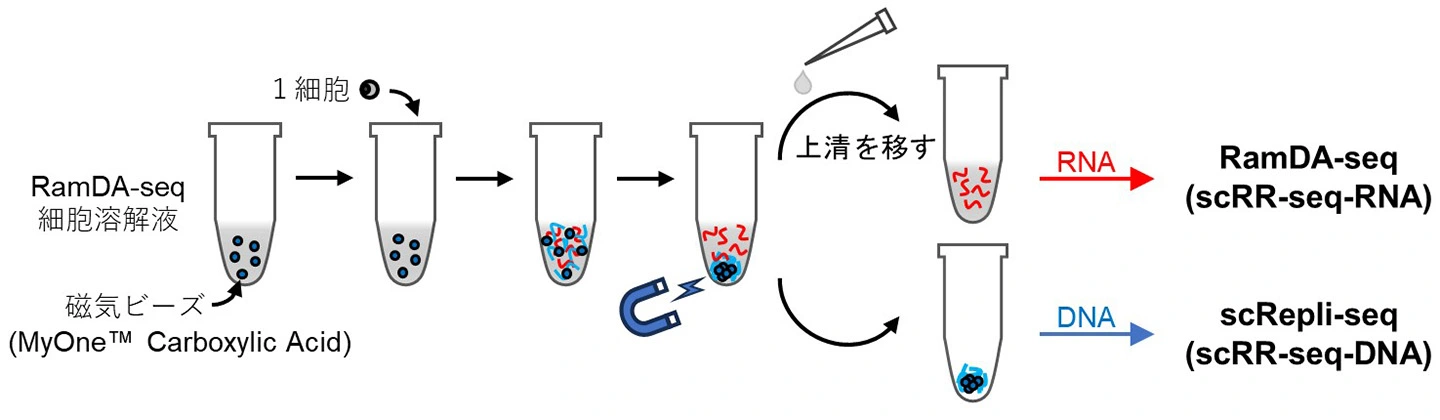
この新しい手法を確立するため、まず1細胞からDNAとRNAを分離する手法を検討しました(図2)。その結果、カルボキシル基を含む磁気ビーズを用いることで、ゲノムDNAを選択的に捕捉できることがわかりました。具体的には、単一細胞を溶解して作ったDNAとRNAを含む溶液中に磁気ビーズを加え、磁気スタンドでビーズに結合したDNAを分離します。これにより、RNAは溶解液中に残り、DNAはビーズに保持されます。この分離操作後に、DNAはscRepli-seq、RNAはRamDA-seqに供しました。
新技術を評価する際に最も重要なのは、得られるデータの質です。scRR-seqを評価するために、従来のscRepli-seqおよびRamDA-seq単独で行ったデータと比較しました。細胞周期のS期[用語11]の中期にいるヒト網膜色素上皮細胞RPE1をFACS[用語12]により回収し(図3a)、scRR-seqのDNA-seq(以後scRR-seq-DNA)を行い、ゲノム全体で複製に伴うDNAコピー数の変化を調べました(図3b)。コピー数はS期におけるDNA複製の状態(1コピー=未複製、2コピー=複製済み)を反映するため、これをもとにゲノム中で複製のタイミングが早い領域、遅い領域を知ることができます。scRR-seq-DNAにより得られた複製タイミングパターンは従来のscRepli-seqによるものとよく一致していました。
一方、scRR-seqのRNA-seq(以後scRR-seq-RNA)は、RamDA-seqと同等の遺伝子転写産物に対するカバー率を有し(図3c)、RamDA-seq単独で検出される遺伝子の約80%(〜1万6000遺伝子)を検出可能であることがわかりました。また、RamDA-seqと同様にポリA型RNAのみならず非ポリA型RNA[用語13]の検出も可能でした。これらの結果から、本手法が1細胞内の遺伝子転写産物を感度良く、しかも効率的に検出できることが確認されました。
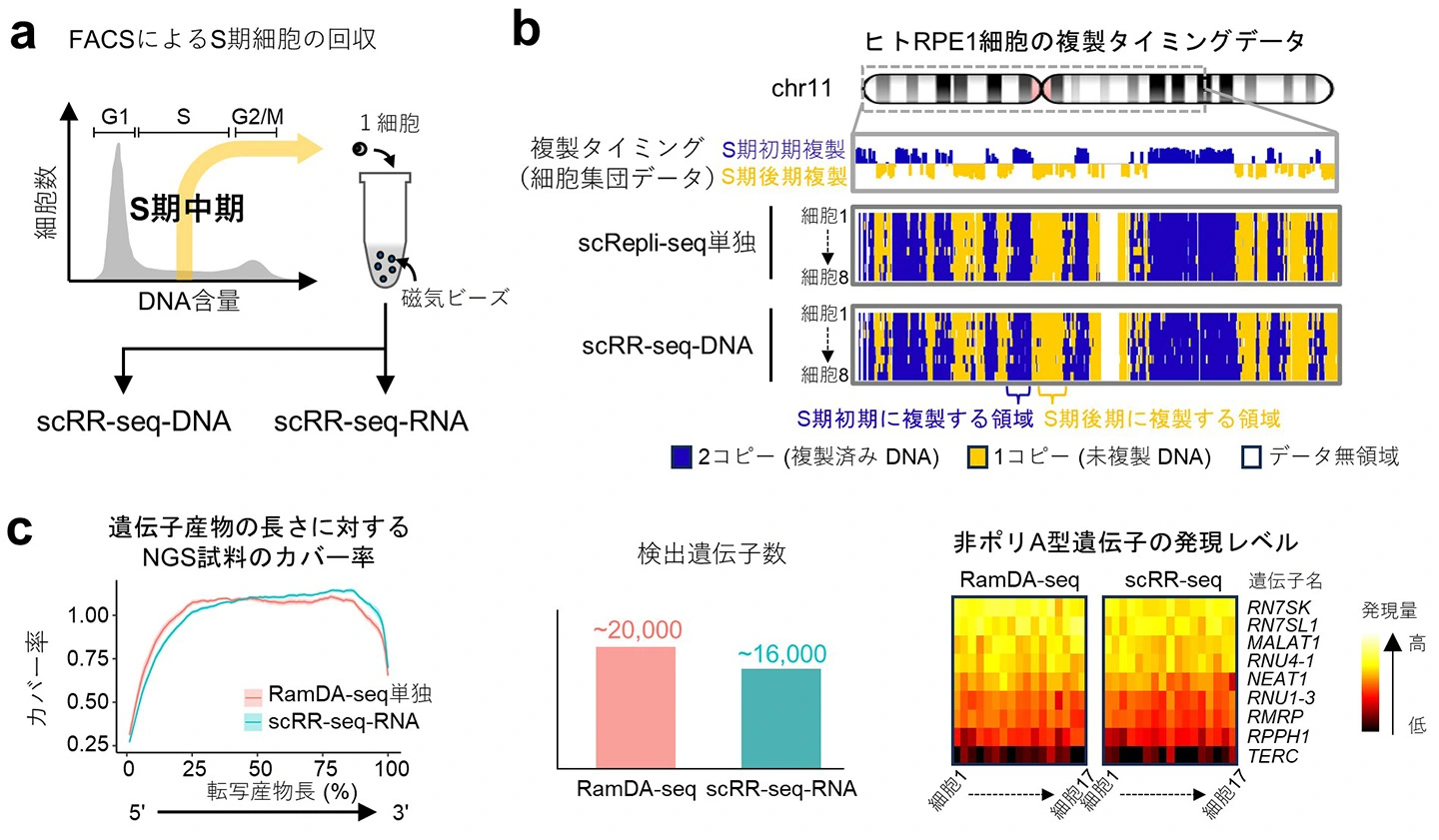
(a)フローサイトメトリー (FACS) により、S期中期のヒトRPE1細胞を一つ一つ分取し、scRR-seqを実施した。得られた結果を、scRepli-seq単独、RamDA-seq単独で得られた結果と比較して評価を行った。
(b) scRR-seqのDNA複製に伴うコピー数変化の結果(scRR-seq-DNA)は、従来のscRepli-seq単独で得られた結果と高い一致を示した。例として、第11染色体(chr11)の結果を提示している。参考として従来の集団細胞解析で得られた結果も併記している。ヒートマップでは、青色で示したゲノム領域はDNAが2コピー存在し、S期の早いタイミングで複製が起こったことを示している。一方、黄色の領域は1コピーのみで未複製のまま残っており、S期の後半に複製されることを示している。
(c) scRR-seqのトランスクリプトームの結果(scRR-seq-RNA)は、遺伝子転写産物の5'末端から3'末端にかけて均一なデータが取得できていることを示しており、これはRamDA-seq単独の結果と同等であった(左)。scRR-seq-RNA はRamDA-seq単独で検出された遺伝子のおよそ80%を同定できたことを示している(中央)。また、scRR-seq-RNA はRamDA-seqと同様に非polyA型RNAも検出可能であった(右)。
以上より、scRR-seqは1細胞から高品質なDNAおよびRNA情報を同時に取得できる強力な技術であり、細胞状態の把握に有用であることが実証されました。
scRR-seq-DNAのコピー数データを用いると、それぞれの細胞が細胞周期のS期のどの段階にあるかを推定できます。これは、2コピーに増えているゲノムDNA領域の割合を算出することでわかります(図4a、右の複製スコア)。この複製スコア順に各細胞のデータを並び替えると、S期を通じたDNA複製の進み具合を詳しく追跡することができます(図4a)。さらに、scRR-seqではDNAだけでなくRNAも同時に調べられるため、S期の進行度に応じた遺伝子発現の変化を追跡でき、これによりS期進行度の目印となる遺伝子を見つけられる可能性があります。実際に、マウス胚性幹細胞(ES細胞)を使って調べたところ、S期の進行度に応じて発現が変わる遺伝子を見つけることができました。これらを「S期進行マーカー」と名づけました(図4b)。これらの遺伝子を分析すると、S期の進み方に沿ったはっきりとした発現の変化が確認できました(図4c)。別の研究で公開されているマウスES細胞のRNA-seqデータにこのマーカーを当てはめてみると、それぞれの細胞がS期のどこにいるかを予測でき、S期進行の様子を再現できました(図4d)。
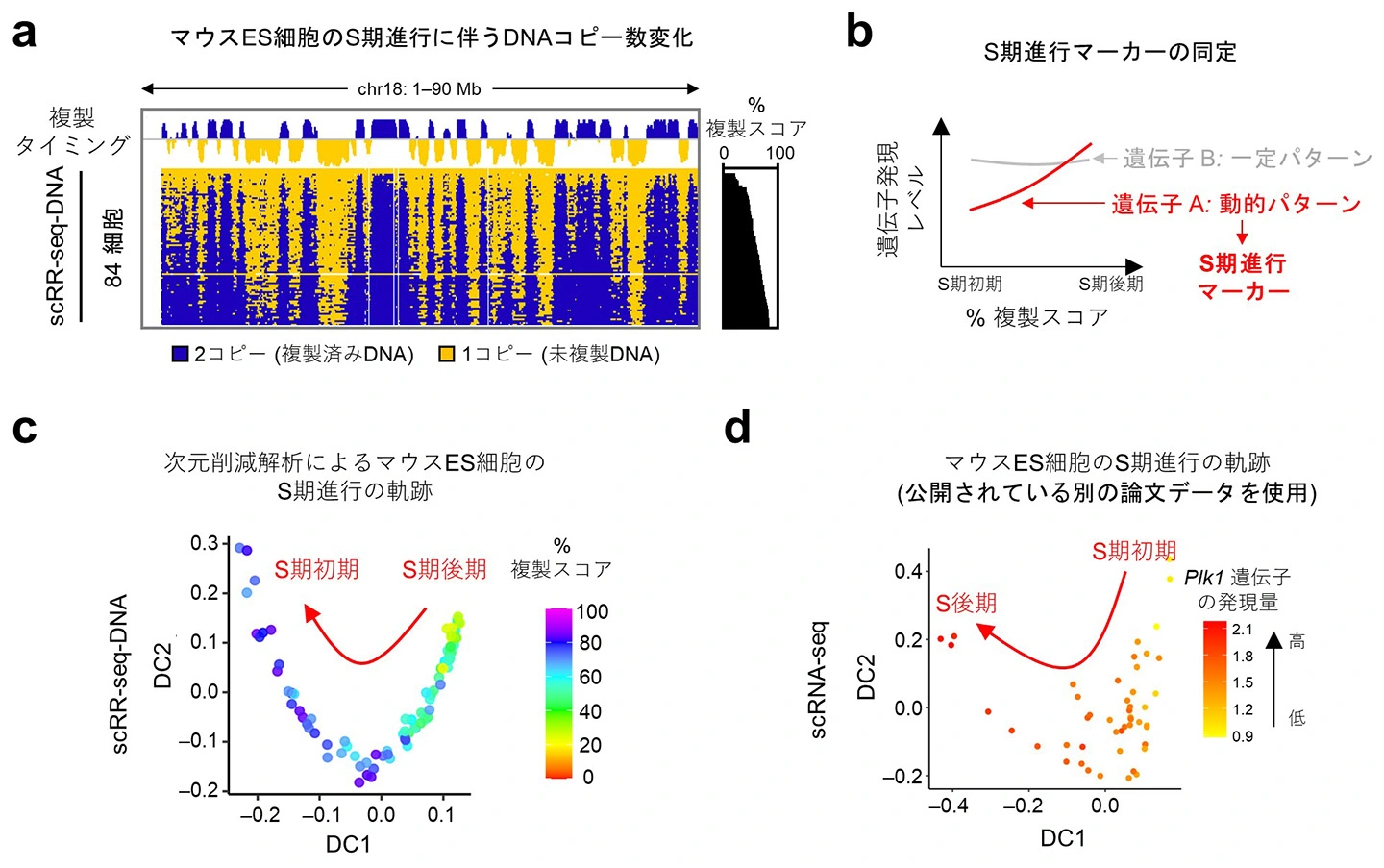
(a) scRR-seq-DNAから得られたCBMS1マウスES細胞の全S期にわたる 複製タイミングのデータ。例として第18染色体(chr18)を示している。コピー数変化のデータを元に算出した細胞ごとのゲノム複製率(複製スコア)を右側に示しており、これにより細胞がS期のどの時期にいるのかを正確に同定できる。
(b) S期の初期から後期にかけて発現量がダイナミックに変化する遺伝子を抽出し、「S期進行マーカー」と定義した。
(c) CBMS1のscRR-seq から得られたマーカーを用いて次元削減解析[用語14]を実施した結果、CBMS1細胞におけるS期進行の軌跡が明らかになった。各細胞(図中の各ドットに相当)は複製スコアに応じて色分けされている。
(d) 新たに同定された S期進行マーカーを別のマウスES細胞株の scRNA-seqデータ(Hayashi et al., Nature Communications 2018)に適用したところ、同様にS期進行の軌跡が観察された。ここでは、S期進行に伴い発現が上昇するPlk1を使用し、Plk1の発現レベルに応じて各細胞を色分けしている。
次に、この技術を用いて、これまでアプローチの難しかった課題に取り組みました。DNA複製の研究分野では、動物細胞の複製に伴うDNAコピー数の変化が遺伝子発現にどのような影響を及ぼすのかが、長年の課題となっていました。細菌では遺伝子発現がDNAコピー数と正に相関しますが、より大きく複雑なゲノムを持つ動物細胞でも同様かどうかは不明でした。そこで、1本の染色体コピーしか持たないヒトHAP1細胞を用いてscRR-seq解析を行いました。複製中のS期細胞に着目すると、一部ゲノム領域は複製済み(2コピー)、他は未複製(1コピー)です(図5a,b)。各遺伝子について、2コピーを持つ細胞で発現が1コピー細胞より高いかどうかを検証しました。その結果、驚くべきことに、多くの遺伝子では複製後に有意な発現増加や減少は認められませんでした(図5b)。このことから、細菌とは異なり、動物細胞では複製に伴うDNAコピー数変化に対して遺伝子発現を安定化させる強固な調節機構が存在する可能性が示唆されました。
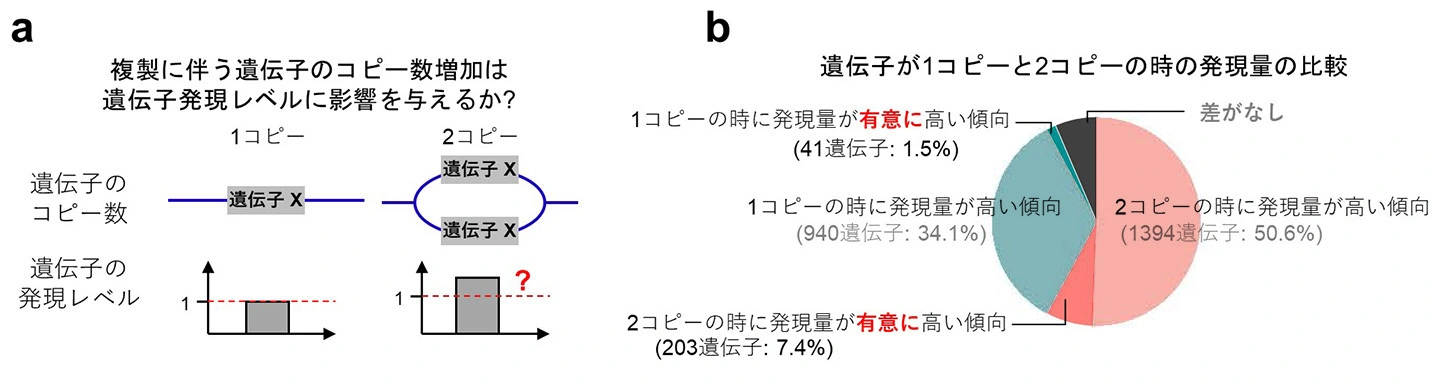
(a)ある遺伝子が複製前の1コピー状態、複製後の2コピー状態にある時、遺伝子の発現レベルに違いがあるのかを調べた。実験は、結果の解釈が容易な半数体HAP1細胞を用いて行った。scRR-seqにより取得した各細胞の遺伝子発現データとDNAコピー数データを用いて、複製に伴うコピー数の増加が遺伝子発現の増加と関連しているかどうかを検証した。
(b) 円グラフは、遺伝子が1コピーの時と2コピーの時の発現パターンを示す。調べた遺伝子のうち7.4%においてのみ、2コピーの時に統計的に有意に発現レベルが高かった。50.6%の遺伝子についても、2コピー遺伝子群でより高い発現を示す傾向が見られたが、その差は統計的に有意ではなかった。
次に、ヒト細胞におけるCNVやゲノム不安定性が遺伝子発現に与える影響を解析しました。正常二倍体線維芽細胞株であるIMR-90細胞を用い、薬剤処理(DNA合成酵素を阻害)することでCNVを誘導しました。scRR-seq-DNAによりG1期[用語15]細胞を調べたところ、一部の細胞でDNAコピー数の増減が確認されました(図6a)。次に、コピー数が変化した領域の遺伝子発現を調べたところ、DNAコピー数の増減が必ずしも遺伝子発現の変動につながるわけではないことが判明しました(図6b)。以上から、DNAコピー数と遺伝子活性の関係は必ずしも連動するものではなく、1つの細胞でDNAとRNAを同時に解析することが重要であることが明らかになりました。
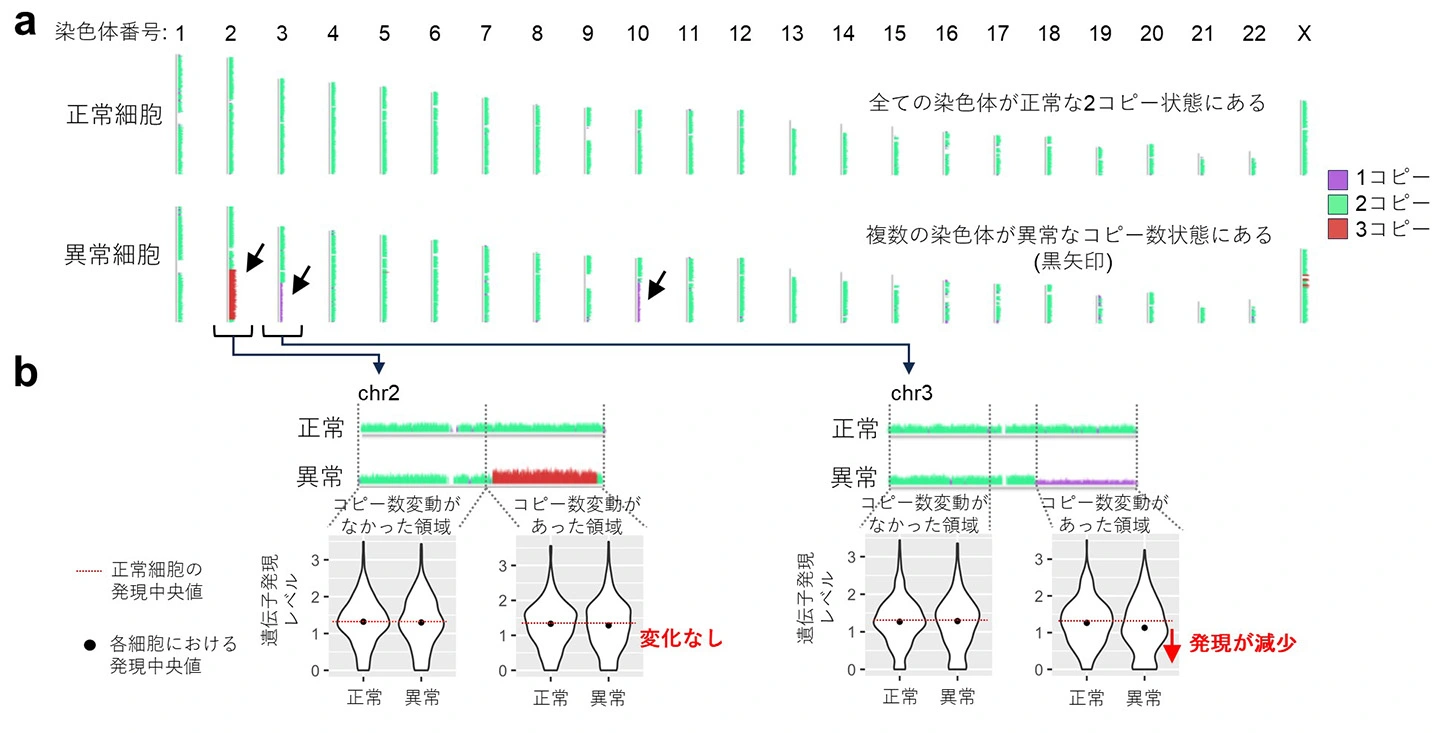
(a) scRR-seq により得られた薬剤処理ヒトIMR-90 細胞の核型。各染色体における DNAコピー数の状態を色で区別している。染色体番号は上部に表示。核型が正常な細胞ではゲノム全体にわたり均一な2コピー状態を示しているが、異常細胞では、大規模な染色体コピー数の変化が観察される(黒矢印)。
(b) コピー数異常を有する2つの染色体の例を示す。下の図は、染色体の各領域に含まれる遺伝子の発現レベルを表している。黒点は各細胞における発現レベルの中央値を示し、赤線は正常細胞における中央値を示す。2番染色体(chr2)に3コピー異常 をもつ細胞は、正常細胞と同程度の遺伝子発現中央値を示した(左)。一方、3番染色体(chr3)に1コピー異常をもつ細胞は、正常細胞と比べて遺伝子発現レベルが低下していた(右)。
今後の展開
今回開発したscRR-seqは、幅広い研究分野への応用や将来的な臨床研究への展開が期待されます。例えばがん研究の分野では、腫瘍内に遺伝子発現パターンやCNVの異なる細胞集団が混在し、疾患進展や治療抵抗性に寄与することが知られています。scRR-seqはこうした細胞の不均一性を検出できる強力なツールであり、診断や治療法の開発に貢献すると期待されます。
さらに、scRR-seqは、通常では実験用に十分な細胞数を確保するのが難しい発生初期の胚の研究にも理想的です。DNAのコピー数変化と遺伝子発現がどのように協調しているかを明らかにし、発生異常の起源を理解する手がかりになると期待されます。また、妊娠初期の染色体異常検出にも応用できる可能性があります。
以上より、scRR-seqは基礎生物学から臨床研究に至るまで幅広く応用可能な、次世代の1細胞DNA/RNA統合解析技術として大きな可能性を秘めています。
付記
本研究は、以下の助成を受けて実施されました。
科学技術振興機構(JST)戦略的創造研究推進事業CRESTの研究領域「ゲノムスケールのDNA設計・合成による細胞制御技術の創出(研究総括:塩見春彦)」の研究課題「潜在的不安定性から読み解くゲノム設計原理(研究代表者:平谷伊智朗、主たる共同研究者:竹林慎一郎、JPMJCR20S5)」、研究領域「[バイオDX]データ駆動・AI駆動を中心としたデジタルトランスフォーメーションによる生命科学研究の革新(研究総括:岡田 康志)」の「ゲノムレジリエンス破綻の理解と未来予測(研究代表者: 二階堂愛、JPMJCR21N6)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「遺伝子の機能的発現を可能にするクロマチンドメインの構築原理解明(研究代表者:竹林慎一郎、JP23K23862)」、科学研究費助成事業挑戦的研究(萌芽)「栄養膜巨細胞に内在するゲノムコピー数調節原理の解明(研究代表者:竹林慎一郎、JP24K21835)」、科学研究費助成事業基盤研究(B)「細胞老化におけるゲノム倍数性変動の多様性とその意義(研究代表者:竹林慎一郎、JP25K02254)」、科学研究費助成事業学術変革領域研究(B)「初期胚が持つ染色体分配異常へのリスクマネジメント(研究分担者:竹林慎一郎、JP25H01445)」、日本学術振興会(JSPS)科学研究費助成事業挑戦的研究(開拓)「1細胞全ゲノム解析の第二世代化と多次元化への挑戦(研究代表者:平谷伊智朗、JP20K20582)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究(A)「マウス初期発生における体細胞型複製タイミング制御確立過程の解析(研究代表者:平谷伊智朗、JP25H00982)」、高深度オミクス医学研究拠点整備事業(東京科学大学)、学術領域展開ハブ形成プログラム(東京科学大学)
用語説明
- [用語1]
- 1細胞解析技術:多数の細胞をまとめて(平均化して)調べるのではなく、1つ1つの細胞のDNA、RNA、タンパク質などを調べる技術の総称。平均化する解析では見えない個々の細胞の違いを明らかにできる。
- [用語2]
- ゲノムDNA:細胞に含まれる完全なDNAセット。遺伝子と遺伝子をコードしない領域を含み、その生物が持つ全遺伝情報。
- [用語3]
- 次世代シーケンス(NGS):DNA配列を大量かつ高速に読み取る最新技術。
- [用語4]
- 核酸増幅技術:少量のDNA/RNAを大量に増やして解析を可能にする方法。特に1細胞解析で重要。
- [用語5]
- 全ゲノム増幅(WGA):少量のDNA(シングルセルなど)からゲノム全体を増幅する技術。
- [用語6]
- DNAコピー数:細胞内の特定DNA領域や遺伝子のコピーの数。
- [用語7]
- DNAコピー数変異(CNV):DNA領域のコピー数が通常(2コピー)と異なること。重複や欠失として現れる。
- [用語8]
- DNA複製:細胞分裂の際に新しい細胞へ完全な遺伝情報を渡すため、DNAを正確にコピーする過程。
- [用語9]
- 非コードRNA:タンパク質に翻訳されないが、遺伝子発現調節などに重要な機能を持つRNA。
- [用語10]
- 全長RNA:5’末端から3’末端まで完全なRNA。
- [用語11]
- S期:細胞周期のDNA合成期。細胞がDNAをコピーする。
- [用語12]
- FACS:細胞中の特定の物質を蛍光で目印を付け、目印が付いている細胞だけを解析・分取する技術。
- [用語13]
- ポリA型RNAと非ポリA型RNA:尾部に連続するA配列を持つRNA(主にmRNA)と持たないRNA。
- [用語14]
- 次元削減解析:膨大で複雑なデータを主要な情報を保ちながら変数を減らして簡略化する計算手法。ゲノムやシングルセル研究でよく使われる。
- [用語15]
- G1期:細胞周期の最初の段階。DNA複製前に細胞が成長し、準備を行う。
論文情報
- 掲載誌:
- Nature Communications
- タイトル:
- scRepli-RamDA-seq: a multi-omics technology enabling the analysis of gene expression dynamics during S-phase
- 著者:
- Rawin Poonperm*, Taiki Yoneda*(*同等貢献の著者), Taito Imada, Saori Takahashi, Takako Ichinose, Hisashi Miura, Tetsutaro Hayashi, Mariko Kuse, Mika Yoshimura, Koji Nagao, Chikashi Obuse, Itoshi Nikaido**, Ichiro Hiratani**, and Shin-ichiro Takebayashi**(**責任著者)
関連リンク
東京科学大学 総務企画部 広報課
- Tel
- 03-5734-2975
- Fax
- 03-5734-3661
- media@adm.isct.ac.jp