ポイント
- AIツールを用いた設計と生細胞スクリーニングで、抗体配列から機能的細胞内抗体を高効率に設計する手法を開発
- 多様なペプチドやヒストン修飾を標的とする機能的な細胞内抗体を高効率で作製することに成功
- 診断や治療にも応用展開可能な細胞内抗体開発の容易化・低コスト化に期待
概要
東京科学大学(ScienceTokyo) 総合研究院 細胞制御工学研究センターの木村宏教授、生命理工学院 生命理工学系 前島大樹大学院生(博士後期課程3年)、米国コロラド州立大学 化学生物学科 スタセビッチ ティモシー(Timothy J Stasevich)准教授(兼 東京科学大学 総合研究院 細胞制御工学研究センター 特任准教授)、九州大学 生体防御医学研究所 大川恭行教授らの研究チームは、AIツールを用いて抗体配列から細胞内抗体を高効率に設計する手法を開発しました。
細胞内抗体は、生細胞内で発現して機能するように設計された抗体であり、治療や診断、生細胞イメージングなどへの応用が可能です。一方で、安定な構造を取らない場合が多く、開発には長らく制約がありました。
本研究では、AlphaFold2[用語1]とProteinMPNN[用語2]というAIツールと、生細胞スクリーニングを統合したAI駆動型パイプラインによって、機能的な細胞内抗体を迅速かつ効率的に取得する手法を開発しました。このアプローチにより設計した26種の抗体配列のうち、19種を細胞内で機能的な一本鎖可変領域抗体へとして発現させることに成功しました。従来法ではこれら19種のうち18種の発現に成功しておらず、本手法の有効性が明確に示されています。また開発した細胞内抗体には、遺伝子発現制御に働く多様なヒストン修飾[用語3]を標的とするものが多く含まれており、今後の機能解明に有用であると考えられます。
抗体配列データベースが急速に拡充される中で、本手法によって、細胞内抗体の設計が加速されることで、より容易かつ低コストでの開発が可能になりより多くの生命科学研究者が開発に取り組めるようになると期待されます。
本成果は、1月2日(米国東部時間)付の「Science Advances 」誌に掲載されました。
背景
細胞は、タンパク質などの分布や役割が異なるさまざまな分子から構成されています。そうした分子が細胞内で局在する場所を検出するには、その分子に特異的に結合する抗体を用いた免疫染色などの手法が用いられます。しかし免疫染色法では、抗体が細胞内の抗原と反応できるように、細胞をあらかじめ化学固定したうえで細胞膜を透過性にする必要があります。従って、抗体による細胞内分子の検出は、生きた細胞では通常行えません。そのため、遺伝子の導入によって生きた細胞内で抗体を発現させ、機能させる方法が開発されてきました。この細胞内抗体は、診断や治療への応用が期待されています。また、通常の蛍光タンパク質[用語4]との融合では可視化できないタンパク質翻訳後修飾などの解析にも有用です。
天然の抗体(免疫グロブリンG)は、重鎖と軽鎖から構成される比較的大きな(アミノ酸の数が全部で1500個程度の)タンパク質複合体で、細胞の小胞体で重鎖と軽鎖がそれぞれ合成され、ジスルフィド結合[用語5]を介して4量体を形成してから細胞外に分泌されます。細胞内抗体として用いる場合は、重鎖と軽鎖に存在する、抗原に結合する領域のみを結合させた(アミノ酸の数が300個弱の)一本鎖可変領域抗体として発現させることが一般的です(図1)。しかし細胞内抗体は細胞質で合成されるため、ジスルフィド結合が形成されずに、細胞内環境において正しくフォールディングされず、可溶性や安定性の確保が困難な場合が多くあります。そのため、天然の抗体のアミノ酸配列をそのまま用いて一本鎖可変領域抗体として発現させても、90%以上は機能しませんでした。
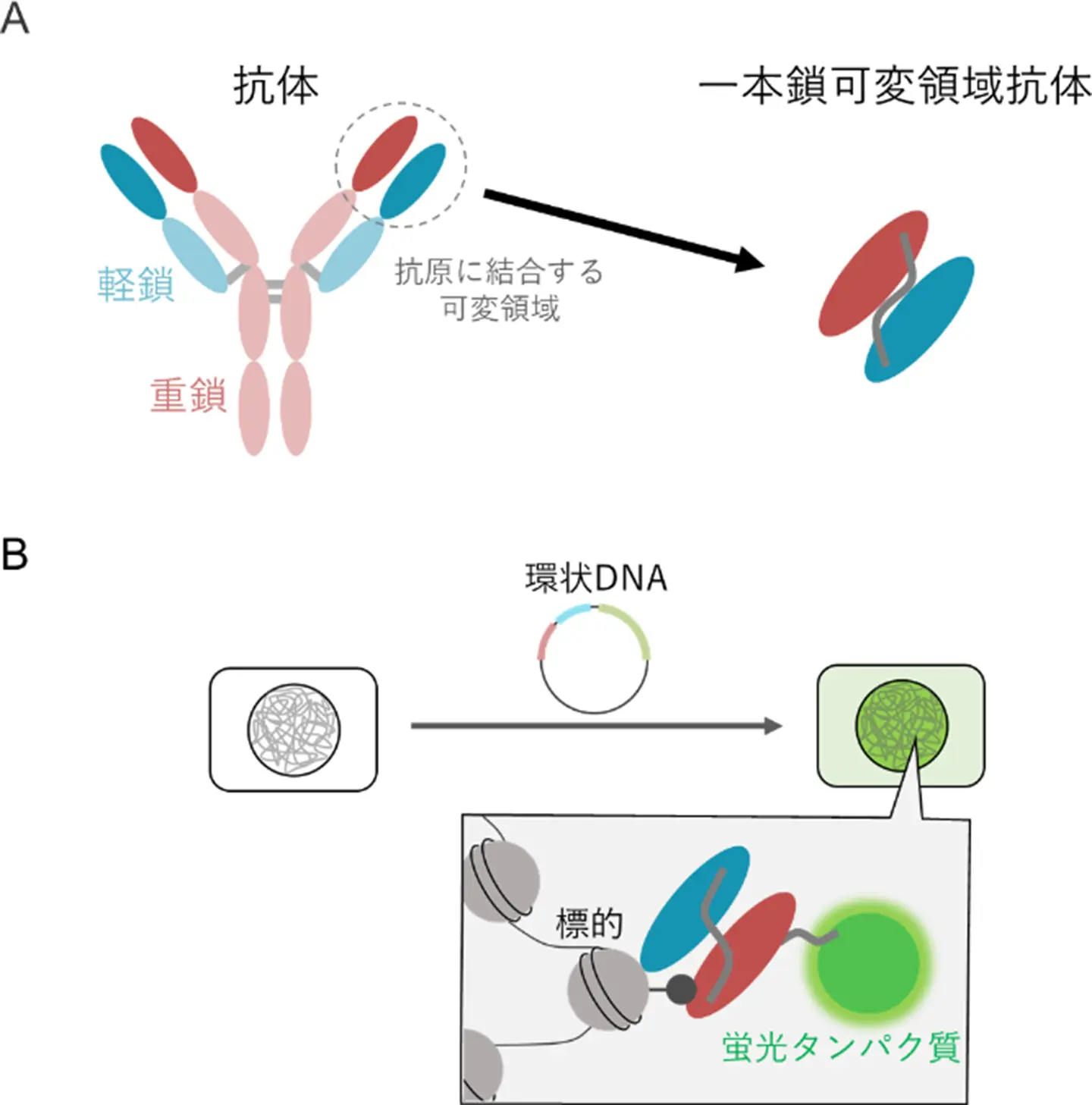
(A)一本鎖可変領域抗体は、重鎖と軽鎖からなる抗体のうち、抗原に結合する領域のみを結合させたものである。
(B)遺伝子の導入により、細胞内で一本鎖可変領域抗体を発現させることで、細胞内の標的を標識することができる。
研究成果
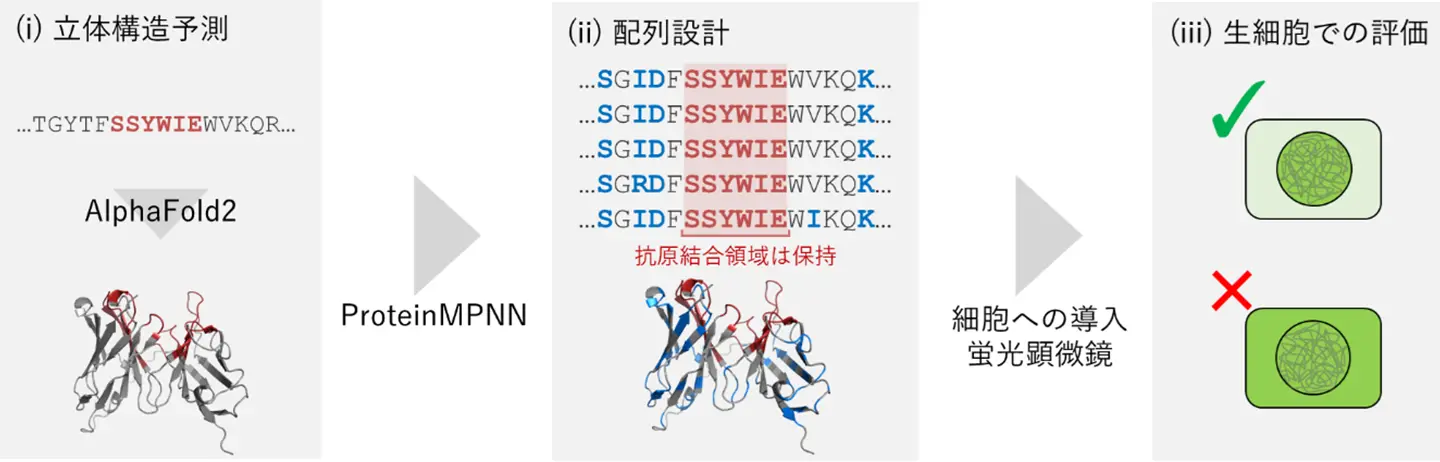
今回の研究では、AIツールを用いた設計と生細胞スクリーニングを統合したパイプラインによって、抗原を認識する領域を保持したまま、抗体のフレームワーク領域を最適化する手法を開発しました(図2)。
具体的には、
- (i)
- タンパク質構造予測AIツール「AlphaFold2」を用いて、一本鎖可変領域抗体の立体構造を予測
- (ii)
- 同じタンパク質構造でも安定性や可溶性がより高いアミノ酸配列を出力するAIツール「ProteinMPNN」を用いて、一本鎖可変領域抗体の抗原結合領域を保持したまま、結合には直接関与しないが構造安定性に寄与するフレームワーク領域を最適化
- (iii)
- ProtenMPNNで予測された配列を5種類選択し、一本鎖可変領域抗体と蛍光タンパク質との融合タンパク質として生細胞で発現させ、局在性を評価する生細胞スクリーニングを実施
という流れで、細胞内で機能的な細胞内抗体候補を選択しました。さらに、それらの候補の性質を細胞内局在や生化学的解析により評価しました。
このアプローチによって設計した26種の抗体のうち、19種について機能的な細胞内抗体を作製することに成功し、約73%の成功率を得ました(図3)。天然の抗体配列由来の一本鎖可変領域抗体がそのまま機能する成功率が10%以下であることから、本設計手法では細胞内抗体の開発効率を飛躍的に向上させることができました。
開発した細胞内抗体には、遺伝子発現制御に重要なヒストン修飾を標的とするものが多く含まれます。
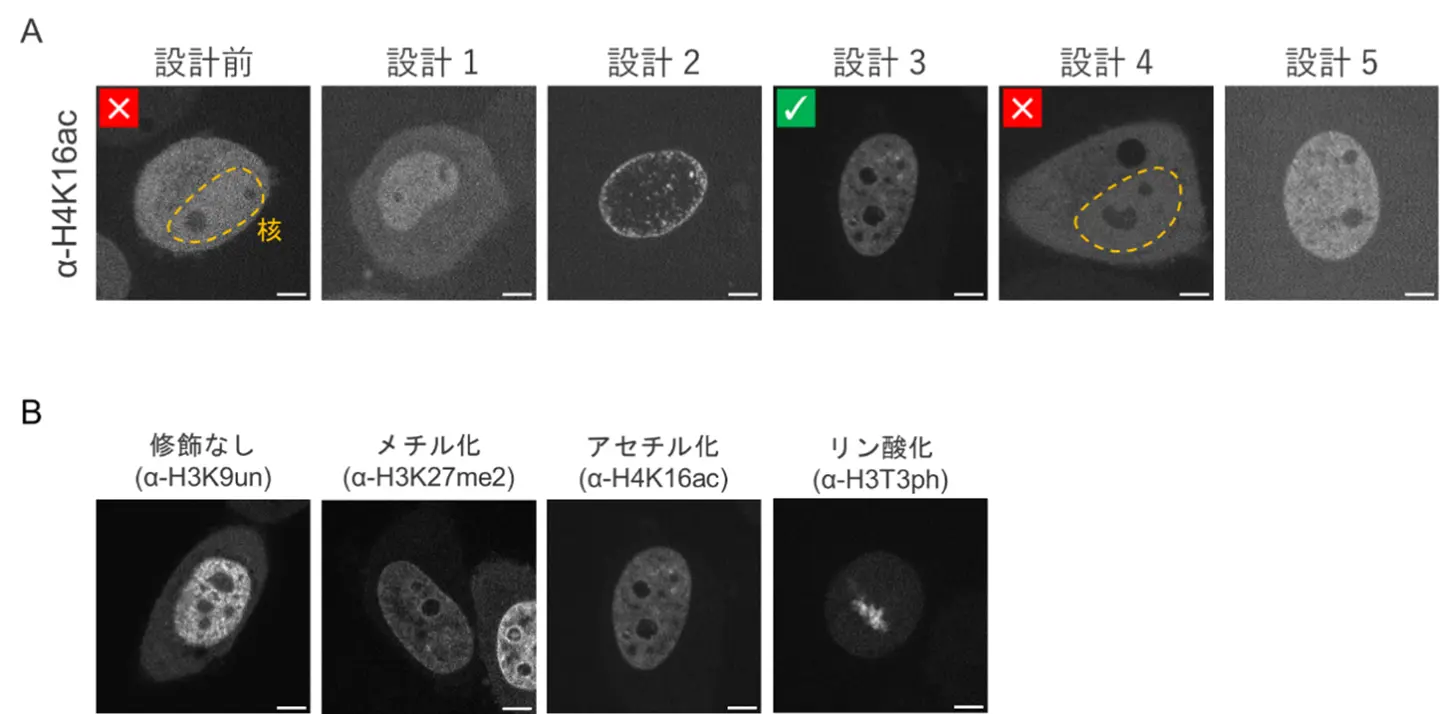
(A) ヒストンH4の16番目のアセチル化リジン残基(H4K16ac)に特異的な抗体を元に作製した細胞内抗体の分布の比較。天然のH4K16ac抗体のアミノ酸配列をそのまま用いた細胞内抗体(設計前)は、標的の存在する核に局在しなかった。一方、本研究の手法を用いて設計した細胞内抗体(設計1〜5)のうち、一部(設計3など)は核に局在した。
(B)さまざまなヒストン修飾(ヒストンメチル化、アセチル化、リン酸化)を標的とする細胞内抗体。スケールバーは5μm。
社会的インパクト
本研究では、AIツールを用いた設計と、生細胞スクリーニングを統合したパイプラインにより、抗体配列から細胞内抗体を高効率に作製できる手法を開発しました。細胞内抗体はその標的となる分子の機能解析に有用であるだけでなく、治療や診断、生体イメージングなどにも広く応用が可能です。抗体を用いた診断や治療に向けた研究が加速する中、抗体データベースの拡充も進んでいます。本手法によって、抗体配列を元にした細胞内抗体の開発が容易かつ低コストで実施できるようになり、広範な生命科学研究者が開発に取り組めるようになると期待されます。
今後の展開
本研究ではAlphaFold2を用いて構造予測を行いましたが、今後、抗原と結合した状態の抗体の構造を予測できるAlphaFold3[用語6]を用いれば、抗原に結合した構造の予測精度が向上し、効率が上がると期待できます。また、今後さらに多くの抗体に対して設計と評価を行い、データを蓄積することによって、細胞内抗体の開発効率をさらに高めることができると考えられます。
付記
本研究は、JSPS科学研究費助成事業(JP17H01417、JP21H04764、JP23H00372、 JP24H02325、JP24H02323、JP24K21949)、AMED BINDS(JP24ama121020、JP24ama121017j0001)、AMED ASPIRE(24jf0126008h0001)、JST CREST(JPMJCR20S6)、JST SPRING(JPMJSP2180)の助成を受けたものです。
用語説明
- [用語1]
- AlphaFold2:Google DeepMind社が開発した、タンパク質の立体構造を高い精度で予測するAIプログラム。2024年ノーベル化学賞の受賞対象となった。
- [用語2]
- ProteinMPNN:AlphaFold2とは逆に、タンパク質立体構造の原子座標からアミノ酸配列を予測するためのMPNN(message passing neural network)というグラフニューラルネットワークを用いたAIプログラム。結晶化されたタンパク質の構造を学習に用いているため、比較的安定な構造を取るアミノ酸を出力する。2024年ノーベル化学賞を受賞したDavid Baker博士らにより開発された。
- [用語3]
- ヒストン修飾:DNAと強く結合するタンパク質であるヒストンの翻訳後修飾であり、部位特異的なメチル化、アセチル化、リン酸化等が存在する。遺伝子発現やDNA損傷修復の制御に働く。
- [用語4]
- 蛍光タンパク質:クラゲやサンゴ等に由来する、蛍光を発するタンパク質。任意のタンパク質と遺伝子レベルで融合させることで、そのタンパク質の生細胞内での局在や動態を検出することができる。
- [用語5]
- ジスルフィド結合:タンパク質中のシステイン側鎖に存在するチオール基(SH)の硫黄原子(S)間の結合。SS結合とも呼ばれる。抗体など細胞外に分泌されるタンパク質は、酸化環境の小胞体で合成されるため、分子内と分子間でジスルフィド結合が形成される。一方、還元環境の細胞質や核ではジスルフィド結合が起こらない。
- [用語6]
- AlphaFold3:タンパク質だけでなく、低分子化合物やDNA、RNAなどの分子の構造を高精度で予測できるAIプログラム。ヒストン修飾のようなタンパク質翻訳後修飾を含む抗原と、抗体が結合した状態の構造も予測できる。
論文情報
- 掲載誌:
- Science Advances
- タイトル:
- AI-assisted protein design to rapidly convert antibody sequences to intrabodies targeting diverse peptides and histone modifications
- 著者:
- Gabriel Galindo, Daiki Maejima, Jacob DeRoo, Scott R. Burlingham, Gretchen Fixen, Tatsuya Morisaki, Hallie P. Febvre, Ryan Hasbrook, Ning Zhao, Soham Ghosh, E. Handly Mayton, Christopher D. Snow, Brian J. Geiss, Yasuyuki Ohkawa, Yuko Sato, Hiroshi Kimura, and Timothy J. Stasevich
研究者プロフィール
木村 宏 Hiroshi Kimura
東京科学大学 総合研究院 細胞制御工学研究センター 教授
研究分野:分子生物学、エピジェネティクス