ポイント
- タンパク質の構造領域と天然変性領域が協調して特定のRNA配列を認識する機構を明らかにしました。
- RNA結合タンパク質FUSがジンクフィンガードメインを用いて特定のRNA配列を認識しながら、隣接する天然変性領域を用いて配列に非依存的にRNAに結合してRNAとの結合親和性を高める仕組みを、分子シミュレーションによって見出しました。
- さらにタンパク質データベースを網羅的に解析し、類似したメカニズムがタンパク質によるRNAやDNA認識において広く存在する可能性を示しました。
概要
東京科学大学(Science Tokyo)生命理工学院 生命理工学系の博士課程学生 木島壮一朗と北尾彰朗教授らの研究チームは、RNA結合タンパク質「FUS」のジンクフィンガードメイン(構造領域)と隣接するRGG2ドメイン(IDR)がRNAの特定の配列を認識する仕組みを、分子シミュレーションを用いて研究しました。
生命現象を包括的に理解するために、細胞内でタンパク質がRNAやDNAなどの核酸の特定の塩基配列をどのように認識しているかを解明することは重要です。核酸を認識するタンパク質の多くは、天然状態で立体構造が安定した「構造領域[用語1]」と柔軟性の高い「天然変性領域(IDR)[用語2]」の両方を含みますが、これらがどのように協調して核酸と結合しているのかは、これまで十分に明らかにされていませんでした。
本研究では、分子シミュレーションを用いてRNA結合タンパク質FUS[用語3]のジンクフィンガードメイン(構造領域)[用語4]とその周辺のIDRがRNAを認識する仕組みを研究しました。その結果、ジンクフィンガードメインは特定のRNA配列を認識して結合しますが、その結合親和性[用語5]はそれほど高くありませんでした。一方、隣接するIDRはRNAの配列に関係なくRNAに結合することで協調的に結合親和性の向上に寄与していました。さらに、IDRを含む他の多数のタンパク質の配列解析から、同様の協調的結合機構が広く存在する可能性も示されました。
この成果は、タンパク質と核酸の相互作用に関する分子基盤の理解を深め、今後の創薬研究や人工分子設計、タンパク質機能の制御技術の開発に向けた重要な足がかりとなると期待されます。
本成果は、7月22日付の「Journal of Chemical Information and Modeling」誌に掲載されました。
背景
生命現象を包括的に理解するには、生命の最小単位である細胞の機能解明が不可欠です。細胞内には、タンパク質、DNA、RNA、脂質など多様な生体分子が高密度に共存しています。なかでもタンパク質は、ほぼ全ての生命活動に関与する中心的な分子です。
多くのタンパク質は、特定の相手分子(基質)に選択的かつ安定的に結合することで機能を発揮します(基質特異性[用語6])。また核酸認識タンパク質は、生物の持つDNAやRNAなどの核酸の特定の塩基配列を認識することで、それぞれのタンパク質に応じた特定の働きを開始します。そのため、タンパク質の核酸の配列認識機構の解明は、生命現象の分子レベルでの理解に直結します。
核酸認識タンパク質の多くは、構造の安定した「構造領域」と、柔軟で揺らぎの大きい「天然変性領域(IDR: Intrinsically Disordered Region)」をあわせ持ちます。細胞内ではこれらが協調して機能すると考えられていますが、従来の研究では多くがいずれか一方の領域にのみ着目していました。
研究成果
FUSのアミノ酸変異は前頭側頭型認知症(FTD)や筋萎縮性側索硬化症(ALS)と何らかの関係を持っていると考えられています。
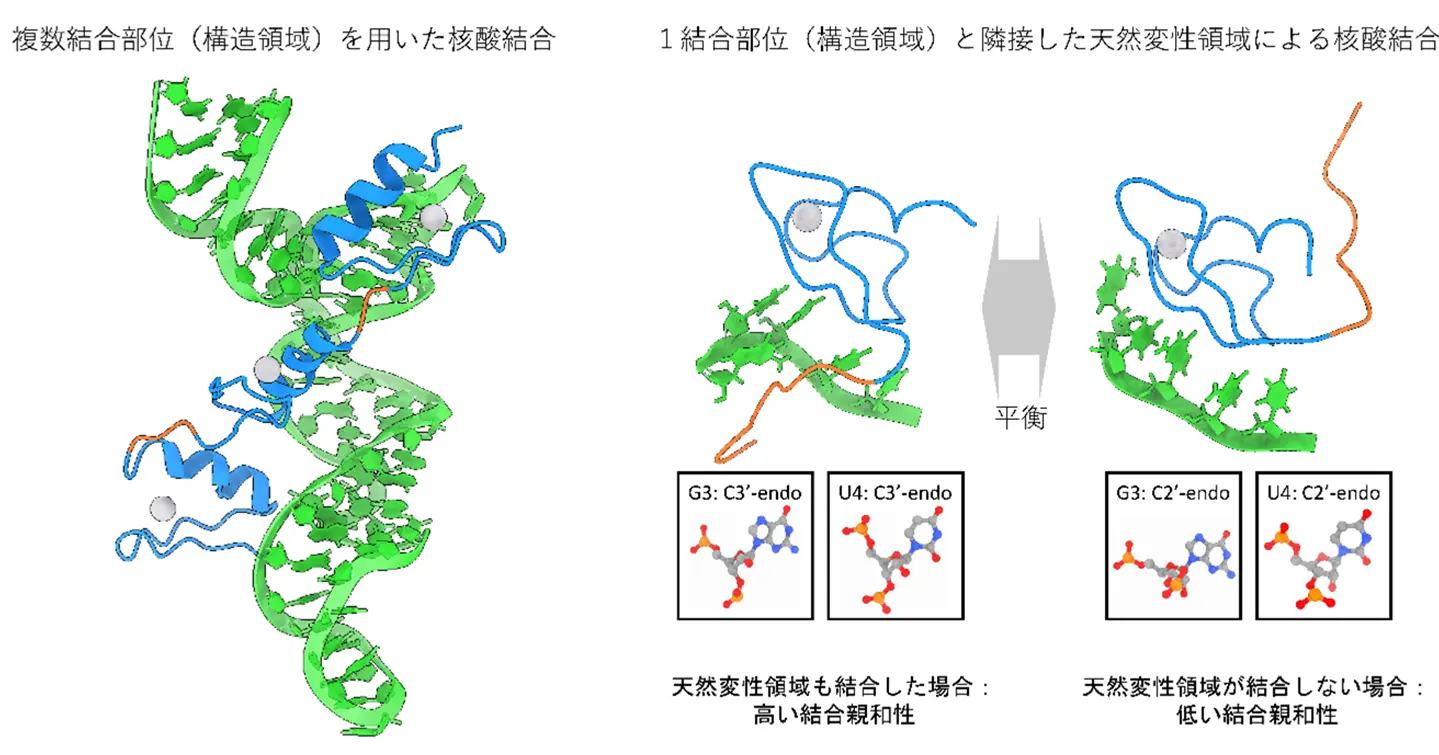
分子シミュレーションの結果、ジンクフィンガードメインが特定のRNA配列と結合する特異性の決定に関与しているものの、その認識するRNA配列はわずか3塩基と短く、結合親和性が低いことが判明しました。一方、ジンクフィンガードメインに隣接する天然変性領域「RGG2ドメイン」は配列に関係なくRNAに結合し、結合親和性を向上させることが明らかとなりました。つまり、構造領域が「何と結合するか(特異性)」を決定し、IDRが「どれだけ強く結合するか(親和性)」を補強するという、協調的な結合メカニズムを持つことを定量的に分子レベルで示しました。
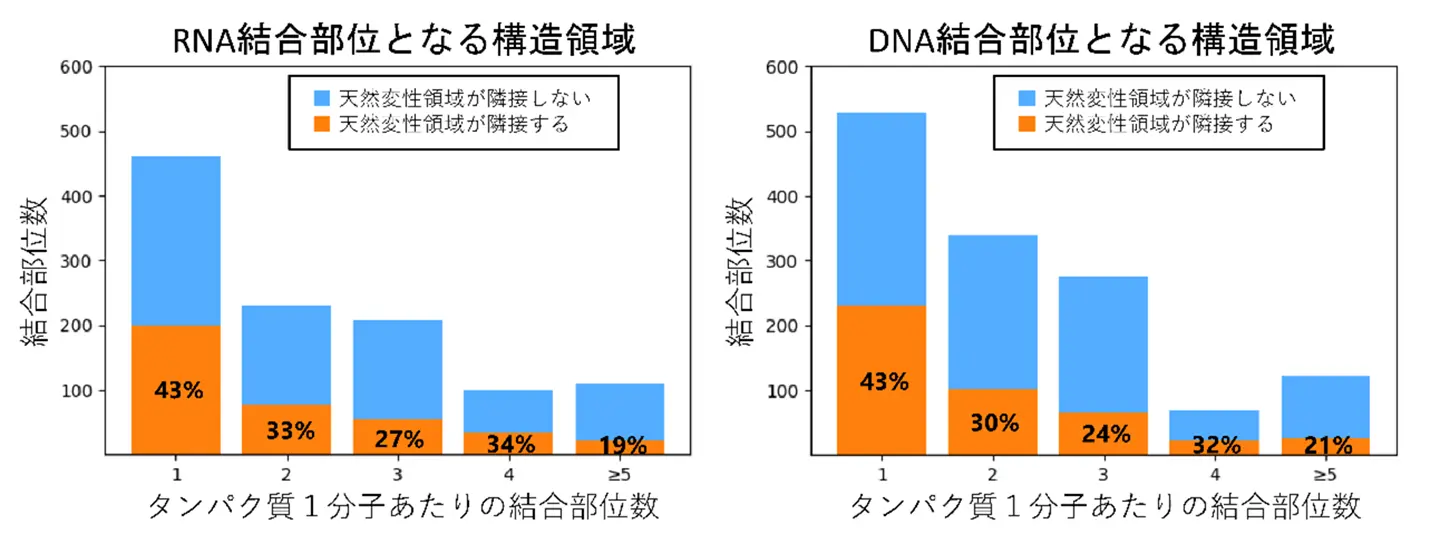
さらに、IDRを含む他のタンパク質群のアミノ酸配列データベースを解析した結果、核酸結合タンパク質は大きく2種類に分類されることが分かりました。1つは複数の構造化した結合部位をもち、結合親和性や特異性を高めるタイプ、もう1つは単一の構造化した結合サイトしか持たないにもかかわらず隣接する天然変性領域を持つことで高い結合親和性と特異性を示すタイプです(図2)。このような役割分担型の結合戦略がFUSに限らず、他の多数のRNA結合タンパク質やDNA結合タンパク質でも広く利用されている可能性があることも示されました。
社会的インパクト
本研究は、タンパク質と核酸の相互作用という生命の根幹を担うプロセスにおいて、構造領域とIDRの協調的機能の分子基盤を明らかにしました。これは、生命現象の統合的理解や、今後の創薬研究、バイオテクノロジー分野での新技術開発にもつながる重要な知見です。
今後の展開
今後は、FUS以外のタンパク質にも同様の結合メカニズムがどれだけ広く存在するかを検証し、協調的結合機構の一般性を評価します。また、実験的検証を通じて、IDRを標的とした創薬戦略の構築や、人工タンパク質設計などへの応用も視野に入れ、分子機械としてのタンパク質の制御技術の確立を目指します。
付記
本研究は、以下の事業・研究領域・研究課題などの支援を受けて行われました。
- 科学研究費助成事業(課題番号:JP23H04058、JP24H02259、JP24H01357)
- 本研究の計算においては、北尾研究室のPCクラスターに加え、東京工業大学TSUBAME4.0、東京大学物性研究所のスーパーコンピュータ(課題番号:2024-Ca-0090、2025-Ca-0038)、自然科学研究機構計算科学研究センターのスーパーコンピュータ(課題番号:24-IMS-C045、25-IMS-C046)、理化学研究所のスーパーコンピュータ「富岳」(課題番号:hp240221、hp250220)の計算資源の提供を受けました。
用語説明
- [用語1]
- 構造領域:天然状態で安定な3次元立体構造をとるタンパク質の領域
- [用語2]
- 天然変性領域(IDR):天然状態で一定の構造をとらず大きく揺らいでいるタンパク質の領域
- [用語3]
- FUS:タンパク質「Fused in Sarcoma」の略称。当初、ヒトの脂肪肉腫など、がんの融合タンパク質として同定されたためこのように名付けられた。RNAと複合体を形成することでさまざまなRNA代謝に関与
- [用語4]
- ジンクフィンガードメイン:亜鉛イオン(ジンク)をつかむ4本の指のような形をしたタンパク質の部分構造(ドメイン)で、核酸に結合するタンパク質によくみられる
- [用語5]
- 結合親和性:分子同士の結合の強さ
- [用語6]
- 基質特異性:タンパク質などの生体分子が、特定の相手のみと安定に結合し、それ以外の相手とは結合しない性質
論文情報
- 掲載誌:
- Journal of Chemical Information and Modeling
- タイトル:
- RNA Binding Mechanism of the FUS Zinc Finger in Concert with Its Flanking Intrinsically Disordered Region
- 著者:
- Soichiro Kijima and Akio Kitao
研究者プロフィール
木島 壮一朗 Soichiro KIJIMA
東京科学大学 生命理工学院 生命理工学系 博士課程2年
研究分野:計算分子生物学
北尾 彰朗 Akio KITAO
東京科学大学 生命理工学院 生命理工学系 教授
研究分野:生物物理学、計算化学、計算生物学
関連リンク
取材申込み
東京科学大学 総務企画部 広報課
- media@adm.isct.ac.jp
- Tel
- 03-5734-2975
- FAX
- 03-5734-3661