ポイント
- 日本人乾癬(かんせん)患者1,415例と対照群3,968例の全ゲノムシークエンス解析を実施し、これまで未解明だった希少変異(rare variant)および構造変異(structural variant)の関与を網羅的に解析。
- 3.3kbの非コード領域欠失(IFNLR1遺伝子座)が乾癬の発症リスクを低下させる保護的効果を持つことを発見。希少変異に基づく遺伝子集約解析により、CERCAMを新規乾癬関連遺伝子として同定し、遺伝子ノックアウトマウス実験で炎症性皮膚病変の増悪を確認。
- 乾癬の分子メカニズムの理解や新たな治療標的探索につながることに期待。
概要
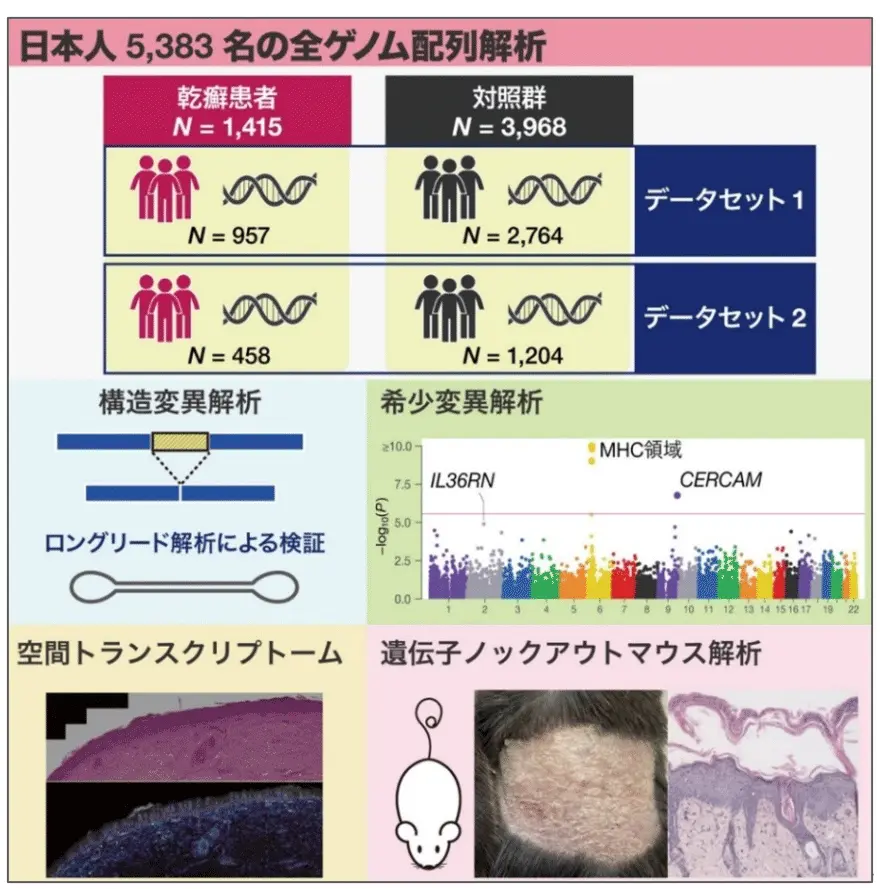
東京科学大学(Science Tokyo) 大学院医歯学総合研究科 免疫学分野の佐藤荘教授、光井雄一助教、東京大学大学院医学系研究科遺伝情報学の曽根原究人助教(研究当時、現:ウェルカム・サンガー研究所Postdoctoral Fellow)、岡田随象教授(兼:大阪大学大学院医学系研究科 遺伝統計学 教授、理化学研究所生命医科学研究センター チームディレクター)、東京大学医科学研究所 附属ヒトゲノム解析センター シークエンス技術開発分野の松田浩一特任教授(兼:同大学大学院新領域創成科学研究科 メディカル情報生命専攻クリニカルシークエンス分野 教授)、名古屋市立大学大学院医学研究科加齢・環境皮膚科学分野の森田明理教授らによる研究グループは、日本人乾癬患者と対照者の計5,383名の全ゲノムシークエンス解析により、これまで未解明だった希少変異[用語1]および構造変異[用語2]の疾患への関与を網羅的に解析しました。構造変異の解析では、IFNLR1遺伝子座の3.3kb欠失が乾癬発症リスクを有意に低下させることを発見しました。希少変異の解析では、CERCAM遺伝子を新たな乾癬関連遺伝子として発見しました。Cercam遺伝子欠損マウスを用いた実験では、乾癬モデルで皮膚炎症の増悪が観察されました。
本成果は、従来のゲノムワイド関連解析(GWAS)[用語3]では見逃されてきた希少変異と構造変異が乾癬の発症に関与することを示すものであり、疾患の分子メカニズム理解や新規治療標的探索につながると期待されます。
本研究は2025年8月22日午前11時(米国東部夏時間)に国際科学誌Cell Genomicsに掲載されました。
背景
乾癬は、皮膚の表皮細胞の過剰な増殖、免疫細胞の浸潤、血管の再構築を特徴とする、代表的な免疫介在性炎症性皮膚疾患です。乾癬は遺伝的要因の関与が大きいことが知られています。これまでに一塩基多型アレイ[用語4]を用いたGWASによって多数の関連遺伝子座が報告されてきましたが、既知の関連遺伝子領域をすべて合わせても説明できる遺伝率は30%未満であり、まだ多くの遺伝的要因が未解明です。その原因の一つとして、従来のGWASでは解析が困難な希少変異や構造変異が十分に調べられていないことが挙げられます。
研究の内容
今回、研究グループは日本全国から集めた1,415例の乾癬患者と3,968例の対照群に対して、大規模な全ゲノムシーケンス(WGS)解析を実施しました。これにより、従来のGWASでは把握が困難だった希少変異や構造変異を含む全ての遺伝的バリアントを網羅的に解析しました。
構造変異解析では、ゲノム全体に存在する22,783の構造変異を同定し、その中からIFNLR1遺伝子座に存在する3.3kbの非コード領域欠失[用語5]が、乾癬に対して有意な保護的効果を持つことを発見しました(図1)。ファインマッピング[用語6]と機能アノテーション[用語7]により、この欠失は皮膚上皮特異的エンハンサー[用語8]を破壊し、IFNLR1発現を低下させるeQTL効果[用語9]を持つことが示されました。
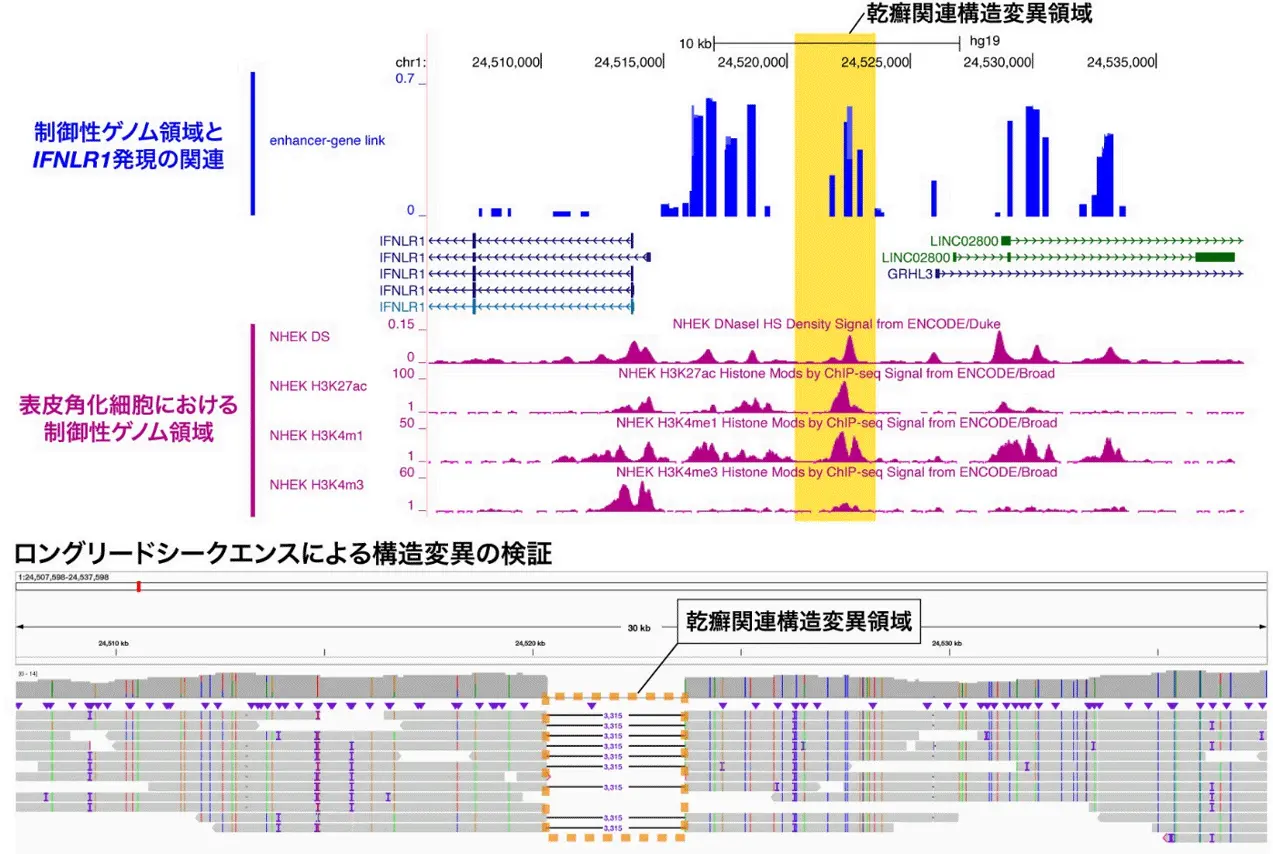
上段の黄色領域および下段の黄色破線枠が本研究で同定された、乾癬に対して保護的に作用する3.3kbの非コード領域欠失を示す。
希少変異については、遺伝子単位の集約解析を行うことで、CERCAMを新規の乾癬関連遺伝子として同定しました(図2)。乾癬患者由来の皮膚検体に対して、空間トランスクリプトーム解析[用語11]を実施したところ、真皮の乳頭層および網状層の線維芽細胞にCERCAMが強く発現することを確認しました。また、IL36RN遺伝子において、希少難治性皮膚疾患である全身性膿疱性乾癬に関連する既知の変異の集積も確認されました。
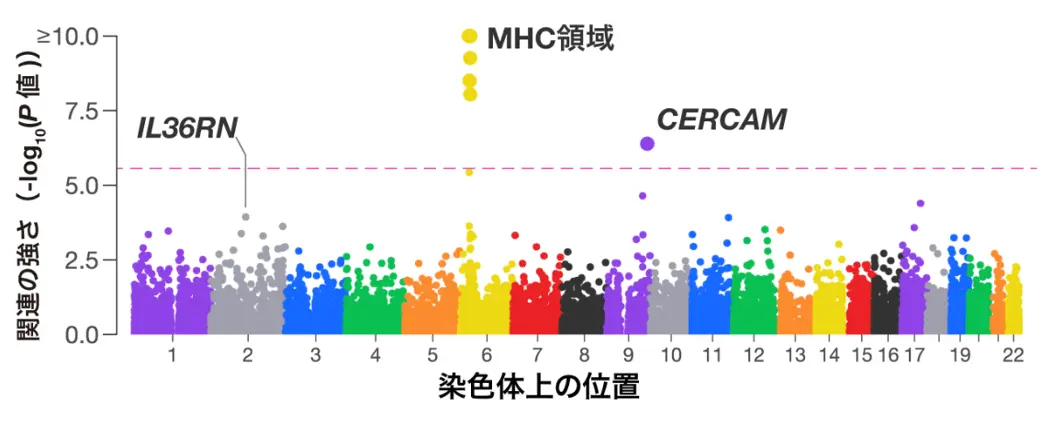
各点は遺伝子の染色体上での位置(x軸)と関連の強さ(y軸)を示す。ピンクの破線はゲノムワイド有意水準を示す。
さらに、in vivo機能検証として、Cercam欠損マウスにイミキモド誘発乾癬モデル[用語12]を適用したところ、野生型マウスに比べて皮膚炎症が悪化し、乾癬関連サイトカイン[用語13]の発現上昇や表皮下領域へのT細胞浸潤増加が観察されました(図3)。
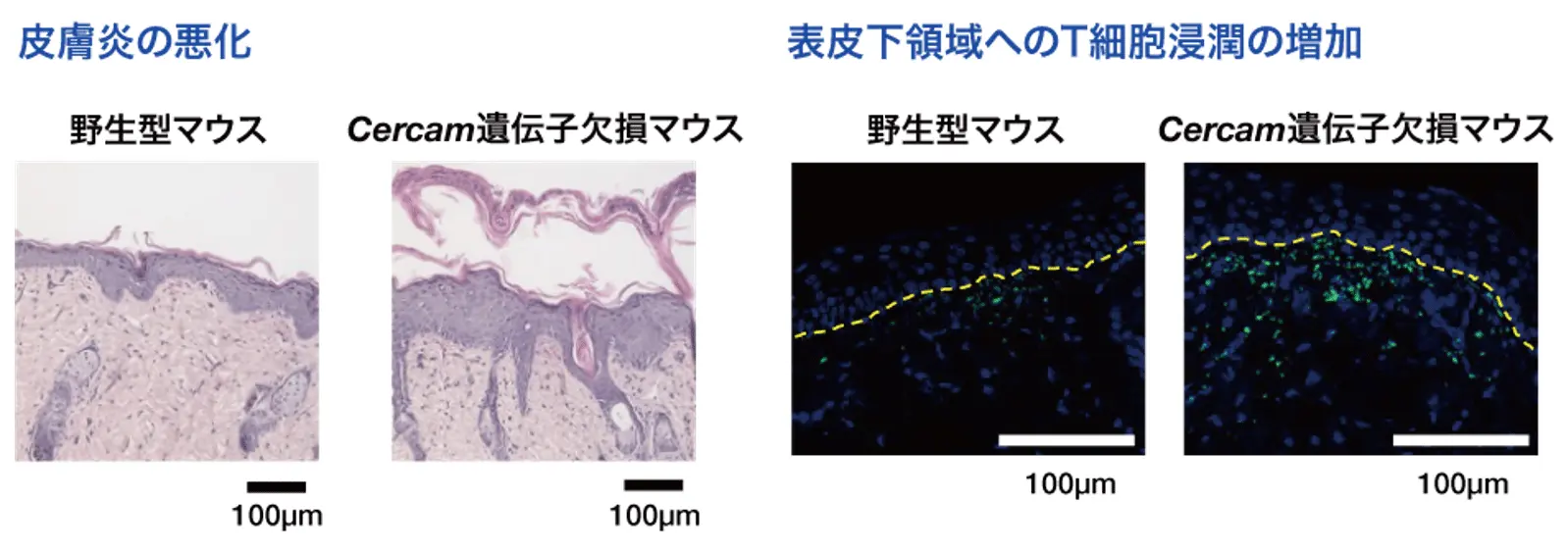
(左):野生型およびCercam遺伝子欠損マウスにおけるイミキモド塗布皮膚の組織像。(右):野生型およびCercam遺伝子欠損マウスにおけるイミキモド塗布皮膚の免疫蛍光染色像。青:DAPI、緑:CD3。黄色の破線は真皮―表皮接合部を示す。
今後の展望
本研究では、日本人集団を対象に希少変異および構造変異の包括的解析を行うことで、乾癬の新規遺伝的要因の同定に成功しました。これらの成果は、乾癬の病態解明に新たな視点をもたらすとともに、個別化医療や新規治療標的の開発に向けた基盤となります。今後は、今回見いだしたIFNLR1遺伝子座の構造変異やCERCAMの希少変異がどのように免疫応答や皮膚バリア機能に影響するのかを分子レベルで解明するとともに、異なる集団や臨床サブタイプ間での比較解析を進めることで、より精緻な遺伝的リスクモデル[用語14]の構築が期待されます。また、これらの遺伝的知見を活かし、より効果的かつ副作用の少ない治療法の開発につなげることが期待されます。
研究助成
本研究は、JSPS 科研費JP23K08850、JP23K08850、国立研究開発法人日本医療研究開発機構(AMED)ゲノム医療実現バイオバンク利活用プログラム(次世代ゲノミクス研究による乾癬の疾患病態解明・個別化医療・創薬)、難治性疾患実用化研究事業(シングルセル解析を活用した肺胞蛋白症の自己抗原特異性解明による治療標的同定)、免疫アレルギー疾患実用化研究事業(全ゲノム・一細胞シークエンス統合解析による関節リウマチの病態層別化と個別化医療実装)、ゲノム創薬基盤推進研究事業(大規模集団ゲノムデータを利用した遺伝子発現制御文法の機械学習による、VUS病原性の網羅的評価と実験検証)、SCARDAワクチン開発のための世界トップレベル研究開発拠点の形成事業「(大規模疾患コホート・アカデミア連携を基盤とするオミックス解析・サーベイランス体制の整備による新興感染症重症化リスク因子の探索)、(ワクチン開発のための世界トップレベル研究開発拠点群 大阪府シナジーキャンパス(大阪大学ワクチン開発拠点))、(遺伝的多様性と機能に関するマルチオミックスを中心としたヒト免疫評価法の確立と支援の為のサポート機関)」、ムーンショット型研究開発等事業(細胞運命転換を用いた若返りによるがんリスク0の世界)、 ゲノム研究を創薬等出口に繋げる研究開発プログラム(ブレインアトラス創生による精神神経疾患のシングルセル・ゲノム創薬)、脳神経科学統合プログラム(生殖細胞・体細胞変異とウイルス配列の多層シークエンス解析による自己免疫性神経疾患のシーズ開発)、革新的先端研究開発支援事業「免疫記憶の理解とその制御に資する医療シーズの創出」研究開発領域「集団・個体・一細胞解像度オミクス解析による免疫記憶の「導入・蓄積・消失」ダイナミクス解明」、国立研究開発法人科学技術振興機構(JST)ムーンショット型研究開発事業 JPMJMS2021、JPMJMS2024、武田科学振興財団、小野薬品がん・免疫・神経研究財団、大阪大学大学院医学系研究科バイオインフォマティクスイニシアティブ、大阪大学先導的学際研究機構(OTRI)、大阪大学感染症総合教育研究拠点(CiDER)、大阪大学ワクチン開発拠点先端モダリティ・DDS 研究センター(CAMaD)、理化学研究所科学研究基盤モデル開発プログラム(TRIP-AGIS)の支援を受けて行われました。
用語説明
- [用語1]
- 希少変異:集団の中で見られる頻度が非常に低い遺伝子変異(例えば0.5%未満)。
- [用語2]
- 構造変異:DNA配列の大きな変化(50塩基以上)で、欠失・挿入・重複・逆位などが含まれる。遺伝子の構造や調節領域へ大きな影響を及ぼしうる。
- [用語3]
- ゲノムワイド関連解析(genome-wide association study: GWAS):遺伝子多型と形質(疾患の有無などを含む、個々人の性質や特徴)との関連を、ゲノム全域にわたって網羅的に探索する解析。現在の一般的なGWASでは、ゲノム全域で数百~数千万に及ぶ遺伝子多型が解析に用いられる。
- [用語4]
- 一塩基多型アレイ:ゲノム上の数十万〜数百万箇所の一塩基多型(SNP)を一度に調べるためのDNAチップ。ゲノムワイド関連解析において広く利用されるが、得られる情報はあらかじめ定まった場所のSNPのみに限定される。
- [用語5]
- 非コード領域:タンパク質をコードしないDNA配列部分。タンパク質の配列自体には影響しないものの、遺伝子発現の制御など重要な役割を果たしている。
- [用語6]
- ファインマッピング:疾患との関連が示されたゲノム領域内で、統計的手法を用いて真に原因となる可能性が高い変異を絞り込む解析手法。
- [用語7]
- 機能アノテーション:ゲノム配列や変異に対して、生物学的な機能や役割を注釈することで、分子メカニズムを解釈しやすくする作業。遺伝子発現データやエピゲノム情報などを用いて行われる。
- [用語8]
- エンハンサー:遺伝子の転写を強めるDNA上の調節領域。細胞や組織ごとに異なる場合があり、遺伝子が適切な文脈で適切に働くために重要な役割を担う。
- [用語9]
- eQTL効果:遺伝子変異が遺伝子の発現量を変化させる効果。
- [用語10]
- ロングリードシークエンス:数千〜数万塩基の長いDNA断片を読み取る新世代のシーケンス技術。ゲノム上の構造変異の検出や複雑な領域の解読に有効となる。
- [用語11]
- 空間トランスクリプトーム解析:組織切片上で遺伝子発現を測定し、その位置情報と統合して行う解析手法。細胞の種類や遺伝子の働きをその空間局在とともに理解でき、組織構造と分子機能の関係を明らかにするのに用いられる。
- [用語12]
- イミキモド誘発乾癬モデル:免疫刺激薬イミキモドをマウス皮膚に塗布し、乾癬様の炎症反応を再現する動物実験モデル。乾癬の病態研究や治療薬評価に用いられる。
- [用語13]
- サイトカイン:免疫細胞などから分泌され、炎症や細胞間の情報伝達を担うタンパク質。乾癬などの免疫疾患の発症・悪化に深く関与する。
- [用語14]
- 遺伝的リスクモデル:複数の遺伝子変異やその組み合わせを用いて、個人の疾患発症リスクを数値的に予測するモデル。個別化医療の基盤となる。
論文情報
- 掲載誌:
- Cell Genomics
- タイトル:
- Whole-genome sequencing reveals rare and structural variants contributing to psoriasis and identifies CERCAM as a risk gene
- 著者:
- Kyuto Sonehara*, Rei Watanabe, Yutaka Matsumura, Yuichi Mitsui, Yosuke Ogawa, Kaori Odomari, Saori Sakaue, Shinichi Namba, Mariko Komuro, Mio Edamoto, Junya Watanabe, Tomomitsu Hirota, Noriko Arase, Yuumi Nakamura, Kimiko Nakajima, Takashi Okamoto, Rika Nishikawa, Kenichi Yamamoto, Ken Suzuki, Toshihiro Kishikawa, Ryuya Edahiro, Yuya Shirai, Tatsuhiko Naito, Noah Sasa, Yosuke Ishitsuka, Junichi Furuta, Kayo Kunimoto, Ikko Kajihara, Satoshi Fukushima, Hideaki Miyachi, Hiroyuki Matsue, Masahiro Kamata, Mami Momose, Ippei Miyagawa, Hiroaki Tanaka, Masanobu Ueno, Toshinori Bito, Hiroshi Nagai, Tetsuya Ikeda, Tatsuya Horikawa, Atsuko Adachi, Tsukasa Matsubara, Emi Nishida, Biobank Japan Project, Koichi Matsuda, Nobuhiro Shojima, Ikuma Nakagawa, Yoshihide Asano, Shinichi Sato, Shinichi Imafuku, Yayoi Tada, Chikako Nishigori, Masatoshi Jinnin, Hironobu Ihn, Akihiko Asahina, Hidehisa Saeki, Toshimasa Yamauchi, Takashi Kadowaki, Tatsuyoshi Kawamura, Shinji Shimada, Ichiro Katayama, Koichiro Higasa, Emiko Noguchi, Shigetoshi Sano, Yoshiya Tanaka, Fumihiko Matsuda, Atsushi Kumanogoh, Mayumi Tamari, Takashi Satoh, Manabu Fujimoto, Akimichi Morita, Yukinori Okada*
*責任著者
研究者プロフィール
曽根原 究人 Kyuto Sonehara
東京大学 大学院医学系研究科 遺伝情報学
研究当時:助教
現職:ウェルカム・サンガー研究所 Postdoctoral Fellow
岡田 随象 Yukinori Okada
東京大学 大学院医学系研究科 遺伝情報学 教授
兼:大阪大学大学院医学系研究科 遺伝統計学 教授
兼:理化学研究所 生命医科学研究センター システム遺伝学チーム チームディレクター
松田 浩一 Kouichi Matsuda
東京大学 医科学研究所 附属ヒトゲノム解析センター シークエンス技術開発分野 特任教授
兼:同大学大学院新領域創成科学研究科 メディカル情報生命専攻クリニカルシークエンス分野 教授
兼:バイオバンク・ジャパン 代表
佐藤 荘 Takashi Satoh
東京科学大学 大学院医歯学総合研究科 免疫学分野 教授
光井 雄一 Yuichi Mitsui
東京科学大学 大学院医歯学総合研究科 免疫学分野 助教
森田 明理 Akimichi Morita
名古屋市立大学 大学院医学研究科 加齢・環境皮膚科学 教授
関連リンク
取材申し込み
東京大学 大学院医学系研究科 総務チーム
- Tel
- 03-5841-3304
- ishomu@m.u-tokyo.ac.jp
東京科学大学 総務企画部 広報課
- Tel
- 03-5734-2975
- Fax
- 03-5734-3661
- media@adm.isct.ac.jp