ポイント
- 酵素Sir2がタンパク質の脱アセチル化反応を効率的に行う「タンデムアロステリック効果」を解明
- 大規模計算機シミュレーションで、反応サイクルの各段階における構造・安定性・アロステリック効果を明らかに
- 脱アセチル化の仕組みの解明は老化抑制・代謝調節・DNA修復などのより深い理解や創薬につながることを期待
概要
東京科学大学(Science Tokyo) 生命理工学院 生命理工学院系の白臻(バイ・ジェン)博士課程学生、Tran Phuoc Duy(チャン・フ・ズイ)助教、北尾彰朗教授は、酵素Sir2が連続する2段階のアロステリック効果[用語1]、「タンデムアロステリック効果」によって、タンパク質の脱アセチル化反応を効率的に行っていることを明らかにしました。
Sir2はDNAと結合するヒストンタンパク質やがん抑制因子p53などの様々なタンパク質を脱アセチル化するサーチュインという酵素群の一種で、老化抑制、代謝調節、DNA修復などにおいて重要な役割を果たしています。研究グループはPaCS-MD/MSM法[用語2]などの大規模計算機シミュレーションを用いることで、Sir2は基質が結合する前のアポ状態では補酵素[用語3]であるNAD+が結合しにくい構造を取り、アセチル化されたタンパク質が結合するとアロステリック効果によってNAD+が結合しやすくなることを明らかにしました。そして、Sir2は脱アセチル化後に2番目のアロステリック効果によって脱アセチル化されたタンパク質を効率的に排出し、再び酵素反応を行える状態に戻ることも解明しました。つまりSir2は2段階の「タンデムアロステリック効果」によって、反応サイクルを効率化していました。
タンデムアロステリック効果に関わるアミノ酸配列は、ヒトも含めた多くの生物種のサーチュインでよく保存されていることも示されました。このことはタンデムアロステリック効果が、脱アセチル化反応を効率化するために備わったサーチュインに共通する機構であることを示唆しています。サーチュインの脱アセチル化の仕組みを詳しく知ることは、老化抑制、糖や脂質の代謝調節、DNA修復のより深い理解や創薬につながることが期待されます。
本成果は、10月13日付(現地時間)の「Journal of Chemical Information and Modeling」誌にオンラインで掲載されました。
背景
タンパク質は翻訳後修飾[用語4]を受けることで、その機能が調節されます。サーチュインと呼ばれる酵素群は、タンパク質の天然変性領域[用語5]などにある、翻訳後修飾によってアセチル化されたリジンを脱アセチル化する酵素です。サーチュインはDNAと結合するヒストンタンパク質やがん抑制因子p53などの様々なタンパク質を脱アセチル化することで、老化抑制・代謝調節・DNA修復などで重要な役割を果たしています。
研究成果
研究グループは、PaCS-MD/MSMなどの大規模計算機シミュレーションを用いて、代表的なサーチュインであるSir2が382番目のリジンがアセチル化されたp53(KAc382-p53)を脱アセチル化するメカニズムを明らかにしました。
サーチュインは基質となるタンパク質を結合した後、補酵素となるNAD+を結合して脱アセチル化を行います。また、Sir2では、NAD+が結合したときにはCBLという部分がNAD+をふたのように覆うことが知られています。しかし、反応サイクルを迅速に繰り返すために、反応後にNAD+が分解されてできたNAMとOAADPrや脱アセチル化されたp53を速やかに排出する仕組みやCBLがどのような構造を取って、それに寄与しているかはこれまでよく分かっていませんでした。
そこで、研究グループはシミュレーションを使って、Sir2が取り得る立体構造分布、KAc382-p53や脱アセチル化されたp53とSir2の結合安定性やアロステリック効果の伝達経路を詳しく調べました。これにより、Sir2は、CBLが開いてNAD+結合部位が露出し、NAD+が結合できるOpen構造と、CBLがNAD+結合部位を覆う3種類の構造(Close1-3)を取り得ることが分かりました(図1)。Sir2は、他の分子が結合していないアポ状態では主にClose1-3構造を取り、Open構造になることは稀です。しかし、KAc382-p53が結合すると基質結合部位から離れたCBLの構造をアロステリック効果によって変化させてOpen構造のみが安定になり、NAD+の結合を促していました(図2左)。次にNAD+が結合するとCBLがNAD+を覆いClose3構造を取ります。さらにp53が脱アセチル化するとNAMとOAADPrが離れ、p53が残ります。この状態では、再びアロステリック効果によってp53の排出を促すようにSir2がClose1またはClose2構造を取ることが示されました(図2右)。この時、脱アセチル化によってp53のSir2への結合が非常に弱くなることもわかりました。つまりp53はSir2から速やかに排出され、Sir2はアポ状態に戻ると考えられます(図1)。
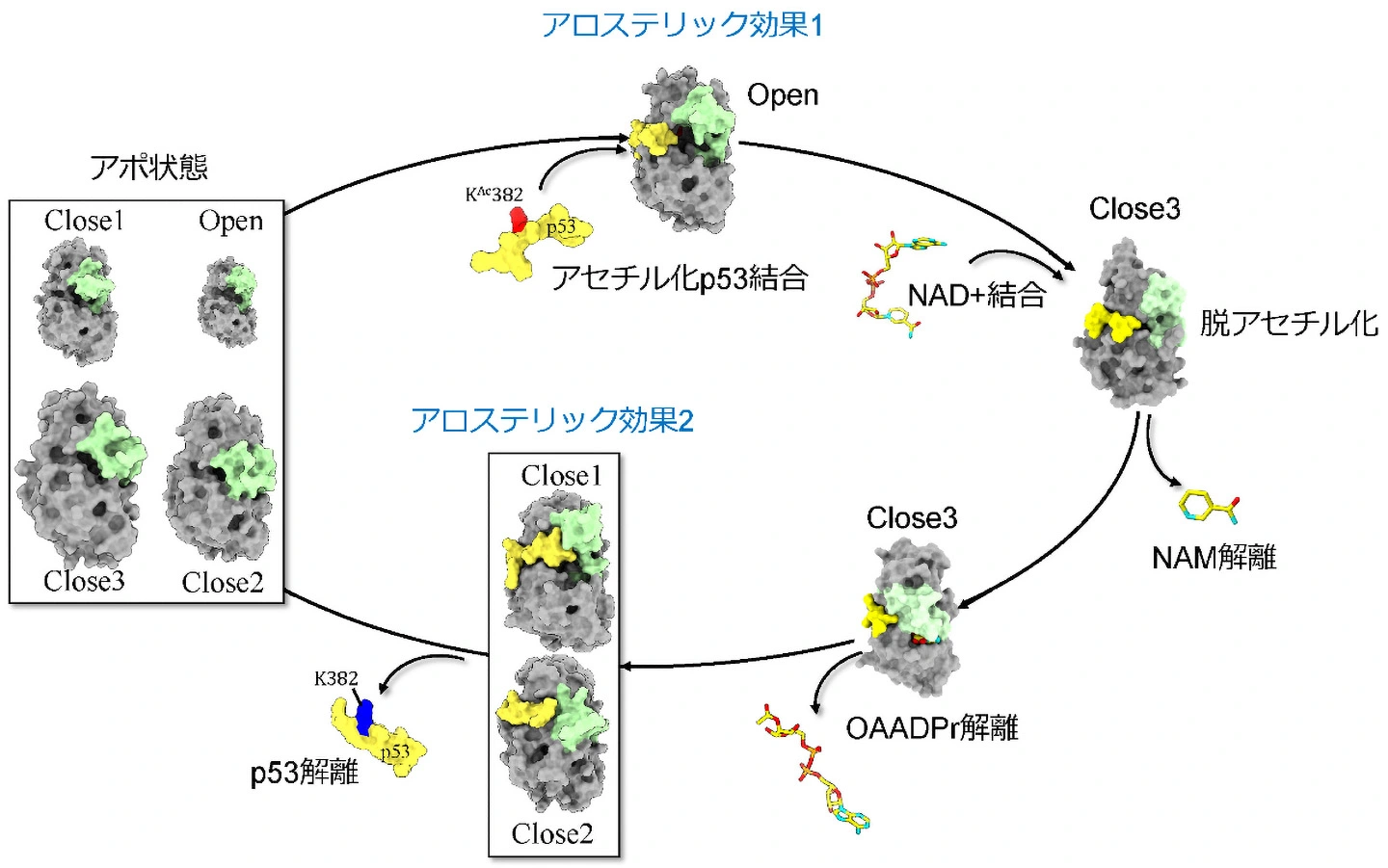
黄緑:Sir2のCBL、灰色:CBL以外のSir2、黄色:p53
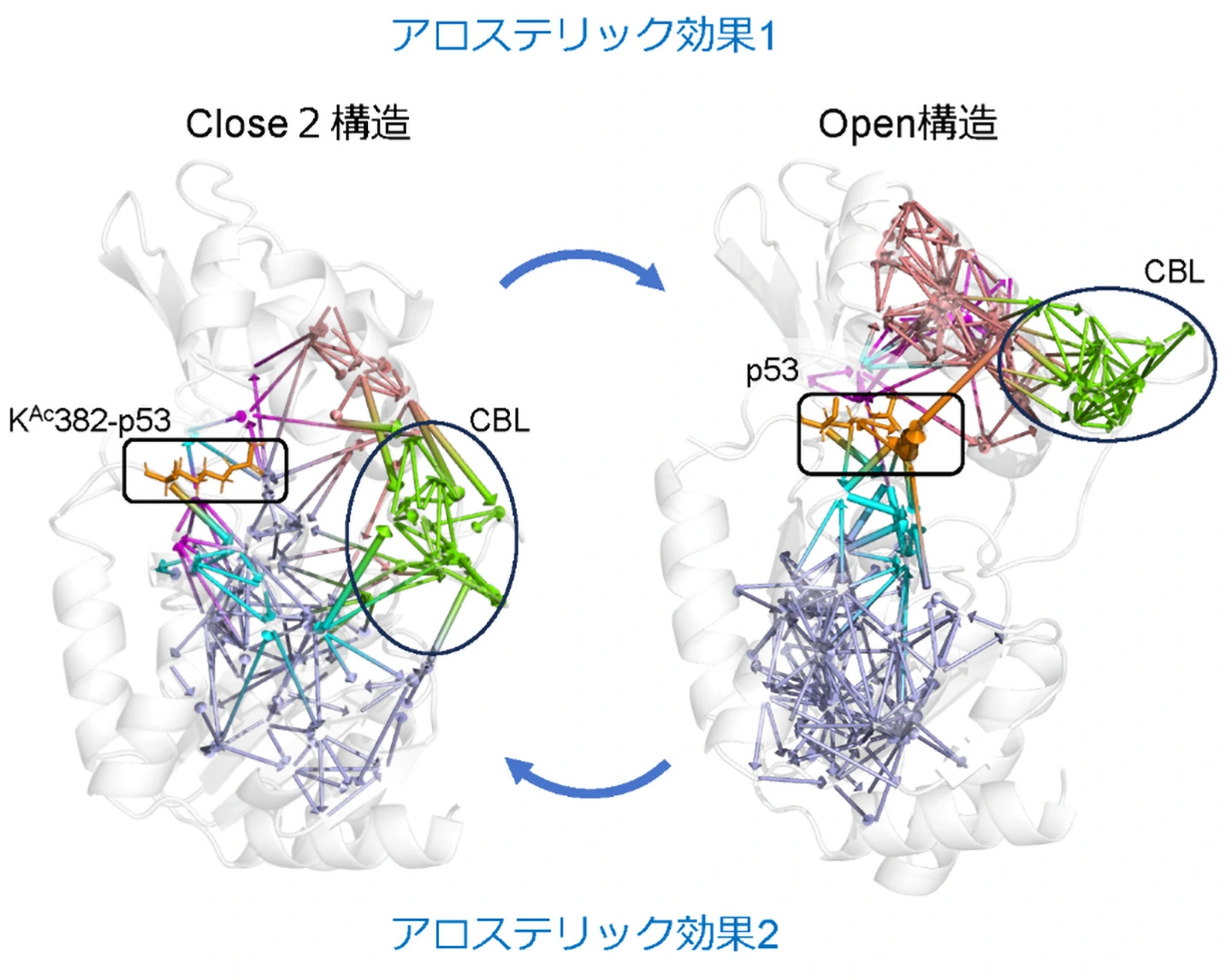
左:基質KAc382-p53(オレンジ)からCBL(黄緑)に至りClose2構造からOpen構造への変化を導くアロステリック効果1と、右:生成物p53からCBLに至りOpen構造からClose2構造への変化を導くアロステリック効果2の情報伝達経路。
このようにSir2は基質が結合する前のアポ状態ではNAD+が結合しにくい構造を取りますが、基質が結合するとアロステリック効果によってNAD+が結合しやすい構造を取ります。脱アセチル化後は次のアロステリック効果によって生成物を効率的に排出し、再び酵素反応を行える状態に戻ります。つまりSir2は、基質と生成物が引き起こす2段階の「タンデムアロステリック効果」によって、反応サイクルを効率化していることが示されました。
社会的インパクト
今回明らかになったアロステリック効果に関わるアミノ酸配列は、ヒトも含めた多くの生物種のサーチュインでよく保存されていることも示されました。このことはタンデムアロステリック効果が、サーチュインが脱アセチル化反応を効率化する共通機構であることを示唆しています。サーチュインによる脱アセチル化の仕組みを詳しく知ることは、老化抑制、糖や脂質の代謝調節、DNA修復などのより深い理解につながります。将来的にはサーチュインの機能を制御する創薬などで、人類の健康増進に寄与する研究の基盤となる可能性のあるインパクトの高い研究です。
今後の展開
北尾研究室では、大規模計算機シミュレーションを用いて脱アセチル化を行う他の酵素、アセチル化などの翻訳後修飾を行う酵素や翻訳後修飾を読み取る分子認識機構の研究を進めています。翻訳後修飾によって変化するタンパク質の機能を、分子間の相互作用という基礎的なレベルで明らかにすることで、ボトムアップ的に翻訳後修飾の生物学的な役割の理解をさらに深化させていきます。
付記
本研究は、以下の事業・研究領域・研究課題などの支援を受けて行われました。
・科学研究費助成事業(課題番号:JP23H04058、JP24H02259、JP24H01357)
本研究の計算においては、北尾研究室のPCクラスターに加え、東京科学大学TSUBAME4.0、東京大学物性研究所のスーパーコンピュータ(課題番号: 2025-Ca-0038)、自然科学研究機構計算科学研究センターのスーパーコンピュータ(課題番号: 25-IMS-C046)、理化学研究所のスーパーコンピュータ「富岳」(課題番号:hp240221、hp250220)の計算資源の提供を受けました。
用語説明
- [用語1]
- アロステリック効果:タンパク質へのある分子の結合が、結合部位から離れたタンパク質の別の部位に影響を与えること。
- [用語2]
- PaCS-MD/MSM法:PaCS-MD(並列カスケード選択分子動力学)とMSM(マルコフ状態モデル)を組み合わせた計算法。分子動力学は、タンパク質・核酸・水・イオンなどからなるシステムが時間に沿って変化していく様子を原子レベルのモデルを使ってコンピュータ上でシミュレーションする方法である。PaCS-MDは、多数の独立な分子動力学を並列で短時間実行し、得られた構造の中から観察したい現象に近い構造を複数選択し、それらを出発構造として分子動力学を再実行するサイクルを繰り返すことで、実時間より数桁短いシミュレーション時間で重要な現象を観察できる。MSMはPaCS-MDで得られた結果を統合して状態の確率や自由エネルギーを計算できる。
- [用語3]
- 補酵素:酵素と結合して反応に寄与する低分子化合物や金属イオン。
- [用語4]
- 翻訳後修飾:タンパク質がリボソームで合成された後、アセチル化・メチル化・ユビキチン化など化学的に修飾されること。
- [用語5]
- 天然変性領域:天然状態で一定の構造をとらず大きく揺らいでいるタンパク質の領域。
論文情報
- 掲載誌:
- Journal of Chemical Information and Modeling
- タイトル:
- Tandem Allosteric Effects of Reactant and Product that Promote Deacetylation Cycles in Sir2
- 著者:
- Zhen Bai, Duy Phuoc Tran, and Akio Kitao
研究者プロフィール
白 臻 Zhen Bai
東京科学大学 生命理工学院 生命理工学系 博士課程3年
研究分野:生物物理学、計算化学
チャン・フ・ズイ Duy Phuoc Tran
東京科学大学 生命理工学院 生命理工学系 助教
研究分野:理論物理学、計算生物物理学
北尾 彰朗 Akio Kitao
東京科学大学 生命理工学院 生命理工学系 教授
研究分野:生物物理学、計算生物学、計算化学